16
2016.06
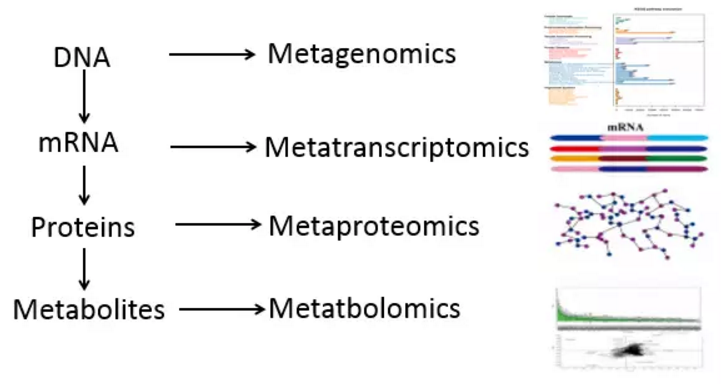
微生物學上炙手可熱的meta研究
Meta - 最早源於電腦科學,是指通過特殊的控制,將一系列動作簡便化進行批次處理的程序。生物學上也有一系列的Meta應用,以獲得宏觀結果為目的,對大量個體研究數據進行統計分析及整合。近年來關於各種 Meta 的研究變得炙手可熱,讓我們來介紹一下吧~
Metadata- 樣品採集時需要蒐集的數據
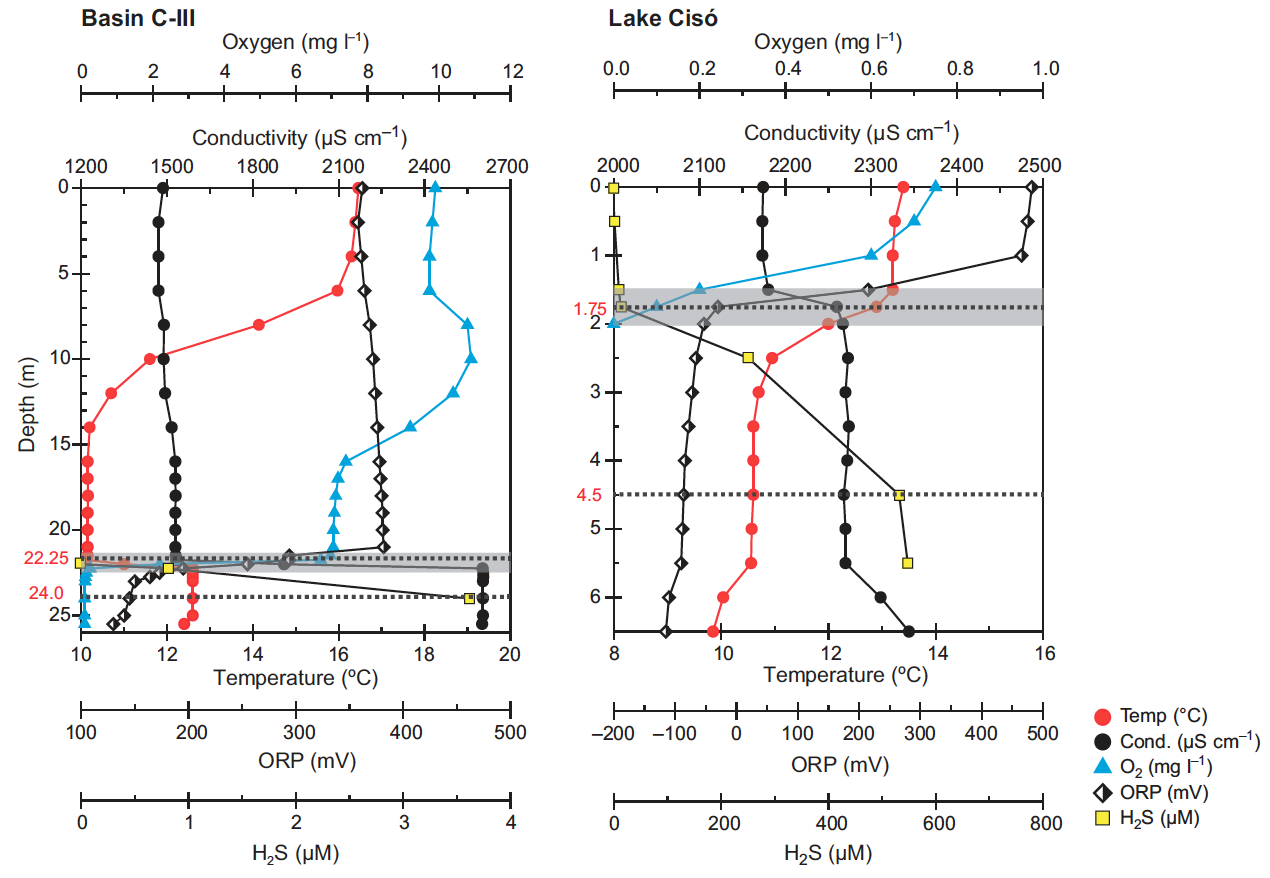
樣品採集是進行後續研究的關鍵步驟,從樣品採集到DNA提取前的每一個操作均應被記錄,包括:採集地資訊(詳細地點、理化性質、周邊環境等)、採集時間、運送條件、保存條件等,樣品採集時蒐集的大量資訊能簡化後續分析並有效地解釋實驗結果。特別是有些需要進行後續環境因子關聯分析的試驗,採樣時的數據收集相當重要!
圖1 Banyoles basin C-III 湖和Cisó湖垂直分佈的理化資訊
Metagenomics (總體基因體/多源基因體)
Metagenomics - “繞過對微生物個體進行分離培養,應用基因體學技術對自然環境中的微生物群落進行研究”(Kevin),是一種以環境樣品中的微生物群體基因體為研究對象,以功能基因篩選或定序分析為研究手段,以微生物多樣性、種群結構、進化關係、功能活性、相互協作關係及與環境之間的關係為研究目的的微生物研究方法。
第二代高通量定序平台具有通量高、準確性高、速度快、資訊全等特點,加快了總體基因體定序推廣應用的速度,在鑑定低豐度的微生物群落,挖掘更多基因資訊方面具有很大優勢,完全能夠滿足常規微生物群體的物種分類研究、群落結構、系統進化、基因功能分析以及物種間的代謝網絡等分析。
儘管總體基因體研究技術比較成熟、應用廣泛,但仍存在一些限制: 1.無法得到微生物基因表現資訊 2.與16S rDNA定序相比,由於需要更高的定序覆蓋度,因此耗時更長,成本更高 3.對DNA數量和品質要求很高,不同實驗室和不同試劑盒提取提取出來的DNA品質差異很大
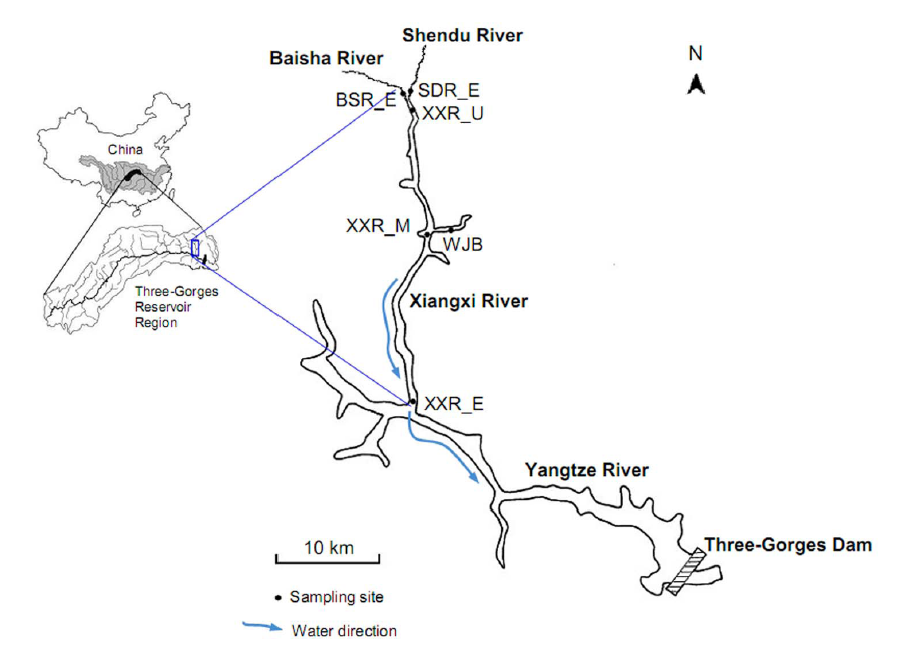
圖2 中科院水生所與諾禾致源合作使用metagenomics研究三峽大壩微生物
Metatranscriptomics (總體轉錄體學)
總體轉錄體定序可用於特定環境下微生物群落中『活躍菌種的組成』以及『活性基因的表現情況』。結合理化因素的檢測,總體轉錄體可研究多樣本間由於理化等指標的差異,時空上不同微生物群落間活躍成分組成的差異分析。
同樣,總體轉錄體研究中也存在一些限制: 1.很難獲取到足夠數量的高品質的RNA用於總體轉錄體研究 2.從數量眾多的rRNA中分離到感興趣的mRNA比較困難 3 .mRNA半衰期比較短,對環境中快速、短暫的關連檢測比較困難 4.關連的資料庫不夠完整
將總體轉錄體與總體基因體定序結合,可研究活躍菌種以及高表現基因的組成情況,揭示特定環境因子影響下菌種的適應性以及基因表現可能的關連機制。
Metaproteomics (總體蛋白質體學)
總體蛋白質體學是一種運用蛋白質技術對特定微生物群落所產生的全部蛋白質進行大規模研究與分析的新技術, 目前總體蛋白體學進行的研究相對較少。但在極端環境中功能基因表達、特殊功能蛋白開發以及生態元素循環等領域顯出強大的功能。
同樣類似於總體轉錄體學,由於資料庫的限制,對所有微生物產生的結構功能複雜的蛋白質進行解析存在很大困難,並且蛋白提取技術的限制、無統一執行標準等因素一直限制著總體蛋白質體學的進步。
Metabolomics (代謝體學)
代謝體學(metabonomics/metabolomics)是繼基因體學和蛋白質體學之後新發展起來的學科,通過對生物體內相對分子質量1000Da以內的小分子代謝物進行定性定量分析,尋找代謝物與生理病理變化的關係。由於能夠更為真實和直觀地反應機體對於刺激的綜合關係,代謝體學已經在疾病研究、藥效檢測等多個領域得到了廣泛應用。
由於一些因素的限制,代謝體學的發展和臨床應用受到限制,主要因素有: 1.資料庫不健全,資料庫中包含的代謝物種類有限 2.由於代謝產物是混合的,很難區分宿主與微生物各自的代謝產物。
參考文獻
- Wang WL , Xu SY , Ren ZG, etal. Application of metagenomics in the human gut microbiome . World J Gastroenterology. 2015, 21(3): 803-814.
- Tomàs LM, Shibu Y, Johannes G, et al. Connecting biodiversity and potential functional role inmodern euxinic environments by microbial metagenomics. Int Soc Micro Ecol. 2015:1-14.
- Yan QY, Bi YH, Deng Y, et al. Impacts of the Three Gorges Dam on microbial structure andpotential function. Sci Rep. 2015, 5: 8605.
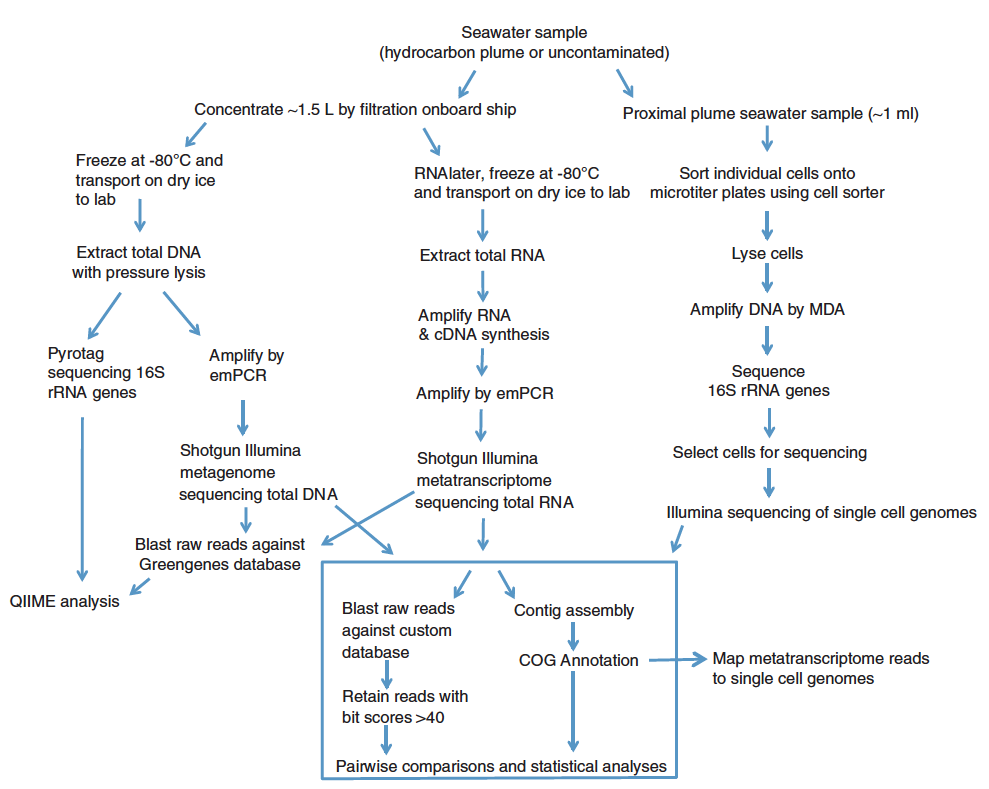
- Olivia UM , Terry CH, Sharon B, et al . Metagenome , metatranscriptome and single-cell sequencing revealmicrobial response to Deepwater Horizon oil spill. Int Soc Micro Ecol. 2012, 6(9): 1715-1727.
- Lee CP, Xiu PC, Poh KK, et al . Non-invasive fecal metabonomic detection of colorectal cancer . Cancer Biol Ther. 2014, 15(4): 389-397.
圖爾思生物科技 / 諾禾致源文案
http://www.toolsbiotech.com/
題圖來源:Vaughn Iverson (華盛頓大學)