17
2016.06
RNA-seq在經費有限下該如何取捨定序深度及樣本量?
研究經費有限,面對提高定序深度或是樣本數,該如何取捨?
採樣非常困難,大量樣本難以獲得,是否就難以得到理想的RNA定序結果?
設置不同定序深度和重複樣本數對差異基因篩選有何影響?
說到RNA定序的實驗方案設計,相信大家或多或少都有上述的困惑。雖然目前定序成本已大幅下滑,但樣本間設置多少重複樣本的問題,仍讓我們面臨超出經費預算的尷尬。那麼在有限的預算下想要盡可能提高手上定序資料的利用價值,我們究竟該選擇定序深度還是樣本數目呢?
刊於Genome Biology的一篇論文:Comprehensive evaluation of differential gene expression analysis methods for RNA-seq data 用數據說話,也許可以為大家解決上面這些疑慮。
研究中構建了一系列down-sample library (大家就簡單理解為隨機採樣的文庫吧),並從每個文庫中隨機抽取50%、40%、30%、20%、10%和5%的reads,運用DESeq, edgeR, limmaVoom和baySeq套件分析結果的交集篩選出共有差異基因,接著作者逐步降低定序深度和重複樣本數,來計算假陽性率和敏感率。
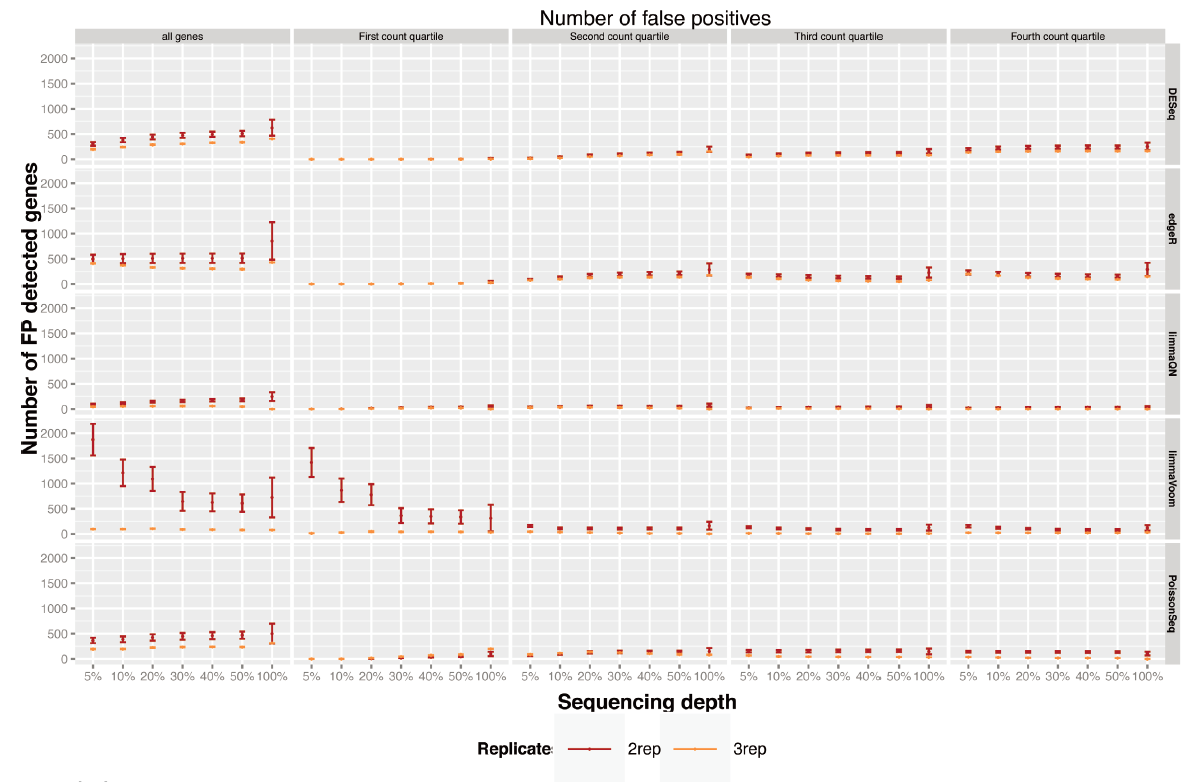
a.假陽性率:
提高定序深度或增加重複樣本數均可降低假陽性率。但是,在25%低位處表現的基因,隨著定序深度和重複樣本數的增加,假陽性率從無到有並逐步遞增;而在75%高位處表現的基因,假陽性率隨著定序深度和重複樣本數的增加卻呈遞減趨勢。
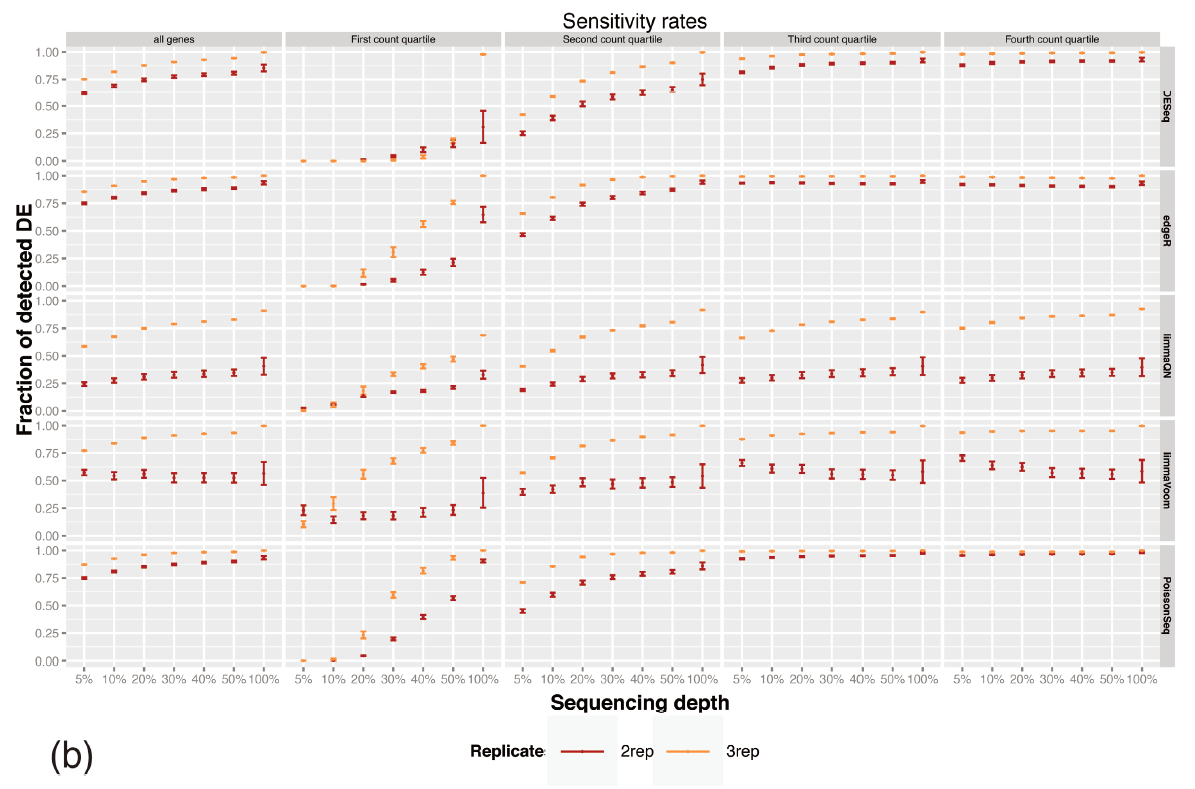
b.敏感率:
在表現量較低的基因中,當抽取5%的reads數和兩個重複樣本時,敏感率不到10%,抽取所有reads和重複樣本時,敏感率達到了100%;而對於表現量較高的基因,提高定序深度和樣本數對敏感率並無顯著影響。總體上,對表現量較高的基因,抽取5%的reads數和兩個生物學重複也能滿足90%的差異基因篩選率。
綜合以上:
(1) 樣本數的選取對差異基因篩選結果的精確性造成的影響比定序深度更大,無論對於技術性重複還是生物學重複都適用。
(2) 對於低現量的基因,reads數和樣本數的選取對差異基因篩選有更大影響,而對於高表現量的基因,提高定序深度對差異基因的篩選幾乎毫無意義。
總的來說,大家要根據自己研究基因的表現量來選取合適的實驗方案:
(a) 對於高表現的基因,低定序深度和少數幾個重複樣本也可以滿足差異基因篩選需求。
(b) 對於低表現基因,提高樣本量能使差異基因篩選極大地受益。
(b) 對於低表現基因,提高樣本量能使差異基因篩選極大地受益。
圖爾思生物科技 / 諾禾致源文案