Eukaryotic RNA-Seq(Quantification)
RNA Sequencing (RNA定序)可以對樣品中所有mRNA做高通量定序及分析,並比較不同實驗條件下轉錄體 (Transcriptome)的變化,找出差異基因,因此RNA-seq / RNAseq 已經逐漸取代以往的Microarray技術成為主流
加入詢價車
分析流程
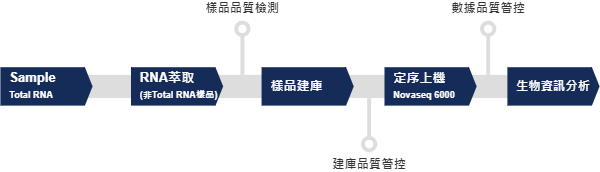
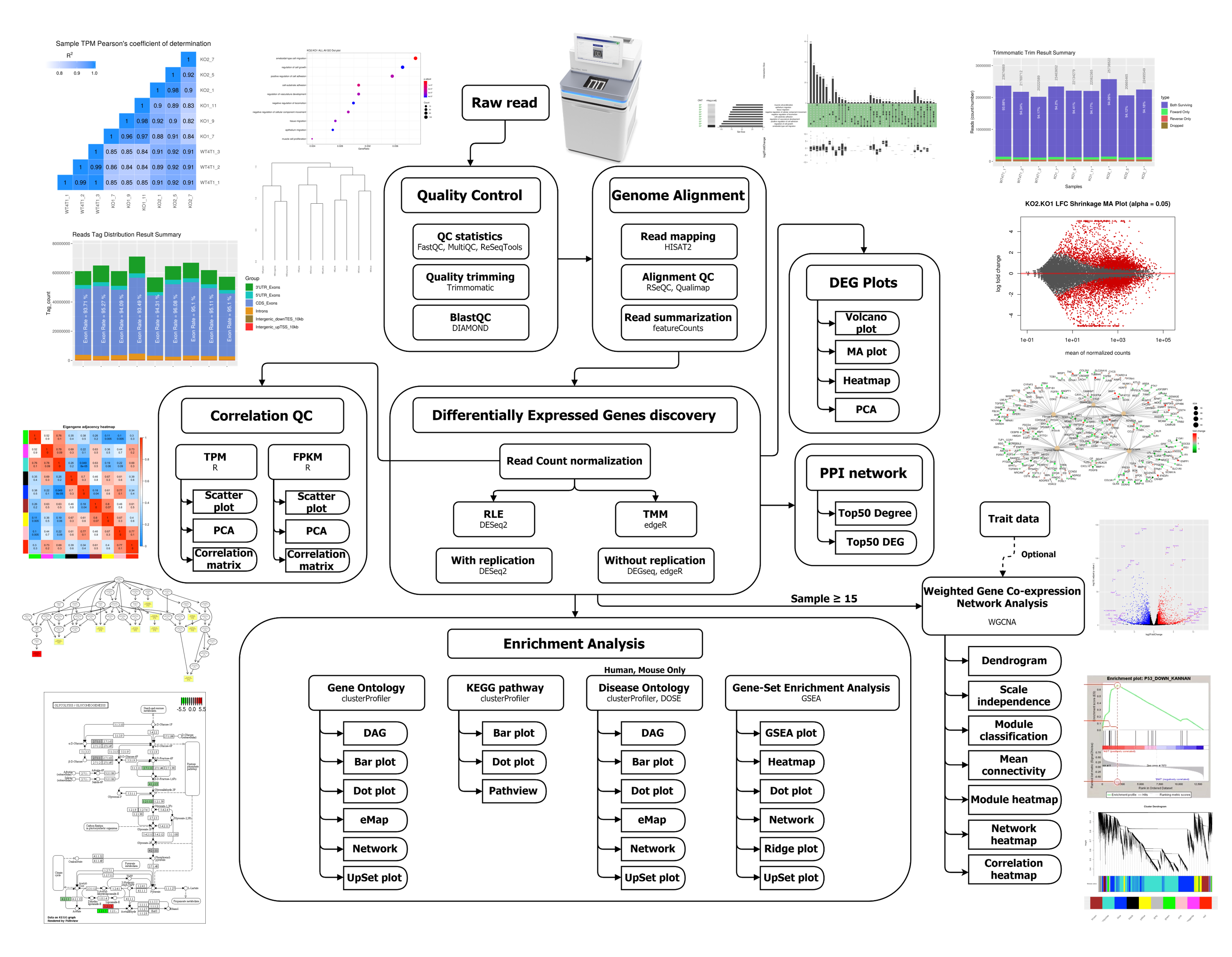
定序完的下機數據 raw reads 以Trimmomatic 執行品質過濾獲得clean reads。Clean reads 以 HISAT2 比對參考序列(如:GRCh38, mm10)並以 featureCounts 計算各個基因表現量 (raw read counts),再透過 RLE / TMM / FPKM 對表現量做歸一化處理。篩選差異基因根據是否有生物學重複區分為:
●具生物性重複樣本,預設以DESeq2,篩選組間基因 |Fold-Change| > 2 及 corrected p-value < 0.05
●不具生物性重複樣本,預設以DEGseq/edgeR,篩選組間基因 |Fold-Change| > 2 及 corrected p-value < 0.005
篩選後以火山圖、熱圖以及主成分分析呈現差異基因結果,並利用 Gene Ontology、KEGG 進行下游的系統生物學分析。除了差異表現基因分析外,流程中亦提供 GSEA 及 WGCNA 兩種進階分析分別做生物調控路徑分析與關鍵基因集探索。
●具生物性重複樣本,預設以DESeq2,篩選組間基因 |Fold-Change| > 2 及 corrected p-value < 0.05
●不具生物性重複樣本,預設以DEGseq/edgeR,篩選組間基因 |Fold-Change| > 2 及 corrected p-value < 0.005
篩選後以火山圖、熱圖以及主成分分析呈現差異基因結果,並利用 Gene Ontology、KEGG 進行下游的系統生物學分析。除了差異表現基因分析外,流程中亦提供 GSEA 及 WGCNA 兩種進階分析分別做生物調控路徑分析與關鍵基因集探索。

樣品需求
1. RNA 樣品:
1. RNA 總量 ≧ 2 μg 2. RNA 濃度 ≧ 50 ng/ μl
3. 樣品體積 ≧ 20 μl
2. 樣品純度及片段完整性: OD260/OD280:1.8 ~ 2.0
OD260/OD230:≧ 2.0
RQN 值:≧ 6.5
建議先行以電泳膠圖確認片段大小及RNA完整度,並於送件時提供膠圖結果
1. RNA 總量 ≧ 2 μg 2. RNA 濃度 ≧ 50 ng/ μl
3. 樣品體積 ≧ 20 μl
2. 樣品純度及片段完整性: OD260/OD280:1.8 ~ 2.0
OD260/OD230:≧ 2.0
RQN 值:≧ 6.5
建議先行以電泳膠圖確認片段大小及RNA完整度,並於送件時提供膠圖結果
定序規格
NovaSeq 6000 , paired-end 150 bp


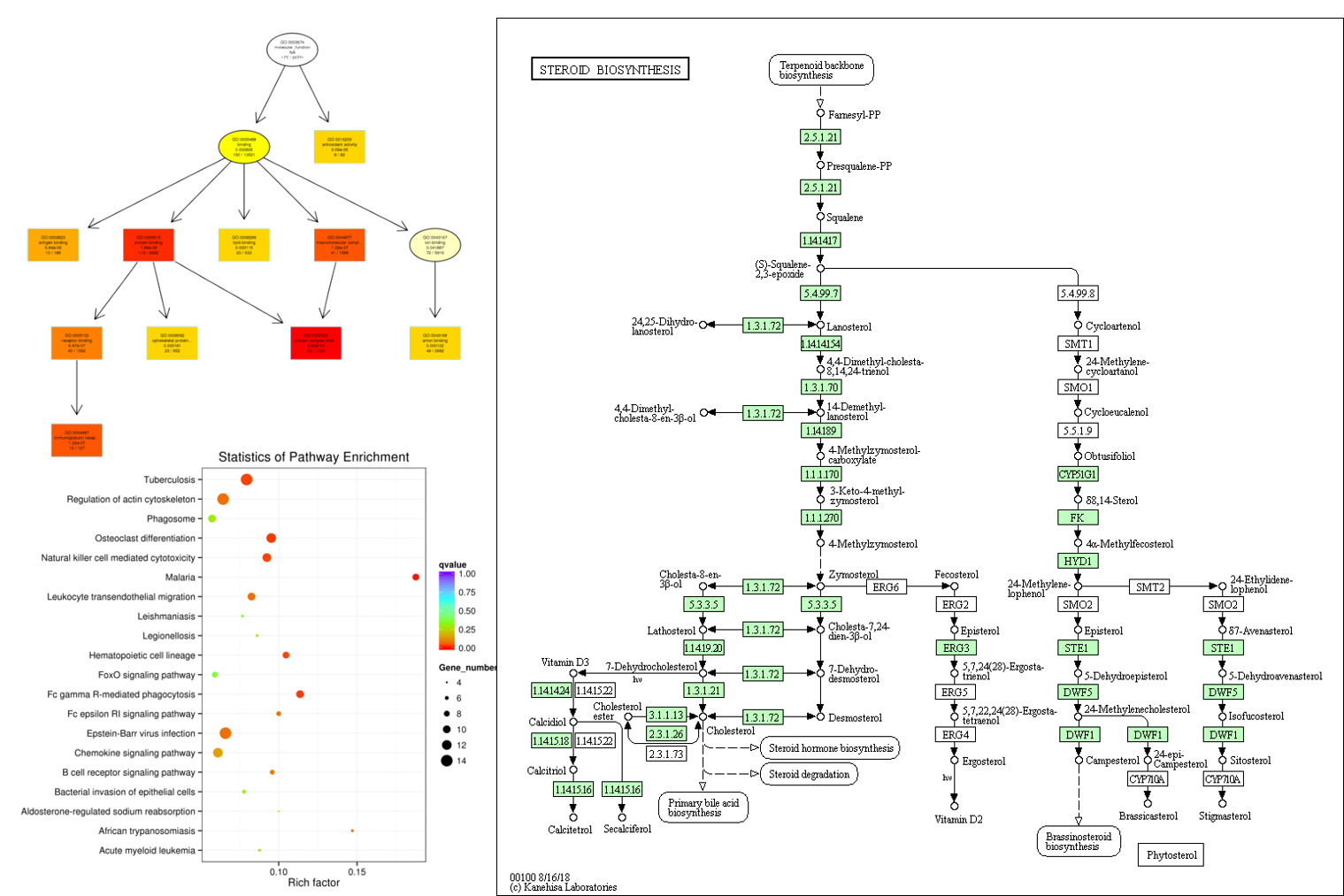
GO&KEGG怎麼看?
藉由RNA-seq可以找出有差異變化的基因表現,但是要如何知道這些基因的功能以及差異基因的關聯是什麼? GO 及 KEGG比對分析就能協助你!
GO
(gene ontology)資料庫,收集的是對各種物種基因功能進行限定和描述的標準詞彙(term),是國際標準化的基因功能描述分類系統。根據基因產物的相關生物學過程( biological_process)、細胞組成 (cellular_component)以及分子功能(molecular_function)三個大類分別給予定義,而每一大類下又包含更多層級具體term,這些定義與具體物種無關。GO
(Kyoto Encyclopedia of Genes and Genomes)是一個綜合資料庫,整合了基因體資訊、化學資訊和生化系統功能資訊,目前包含了16個子資料庫。比如,KEGG PATHWAY資料庫包含了圖解的細胞代謝、膜轉運、訊號傳導等路徑信息; KEGG GENES資料庫、KEGG GENOME資料庫則包含了部分或者完整序列的基因/基因體資訊;KEGG Orthology(KO)是KEGG直系同源資料庫,將各個KEGG註釋系統聯繫在一起,將分子網路和基因體資訊連結起來,根據直系同源關係,實現跨物種的基因體或轉錄體的功能註釋。

分析工具怎麼選?
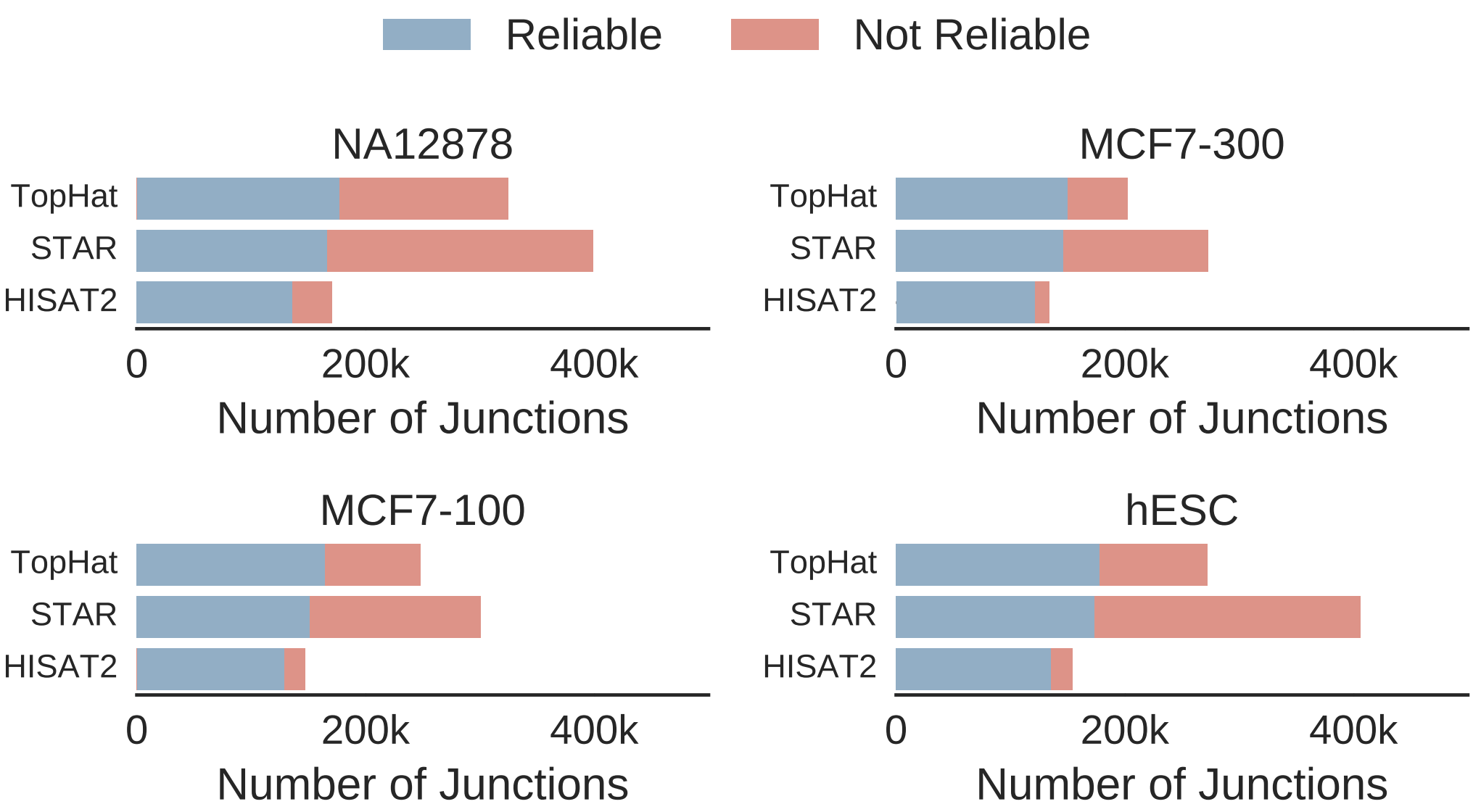
隨著RNA-seq技術的成熟與發展,分析工具也不斷被開發出來並發表,我們整理 2017 年在 Nature Communications 上發表的文章『Gaining comprehensive biological insight into the transcriptome by performing a broad-spectrum RNA-seq analysis』,分別以 short- (illumina) & long-read (PacBio) 定序相同的樣品,比較39種分析工具,檢視各個分析階段的檢測結果,基於結果的準確度與分析時間來評估最佳分析方法、工具與組合流程,並根據文獻測試的結果持續調整升級我們的分析流程,趕快來一探究竟!

採樣建議
樣品建議:
| 血液樣本
新鮮 / 冷藏保存全血:使用 EDTA 採血管收取 5 mL (建議 10 mL)。
Buffy coat:0.5 mL (建議分離自 5-10 mL 全血),分離後以 -80 度保存及運送。
| 動物組織樣本
新鮮或冷凍組織檢體:100 mg (建議 200 mg),-80 度保存及運送。
| 細胞樣本
細胞株樣本:1x10^6~1x10^7 cells,-80度保存及運送。
| 植物組織樣本
新鮮組織或幼苗的嫩葉/嫩芽,取 0.5 g - 1 g 進行萃取,完成 RNA 萃取後送件。
多醣多酚類含量高的植物,後續需要較多的核酸純化步驟,建議取到 4 g 進行萃取,完成 RNA 萃取後送件。
其它注意事項:
- 全血樣品避免使用玻璃材質且需加入 EDTA 抗凝劑,由於溶血作用會嚴重影響 RNA 萃取效率,運送過程的搖晃有高機率造成溶血,建議採血後盡速完成 RBC lysis 處理,或是 Buffy coat 分離,若必須運送請保持 4 度避免搖晃和冷凍。
- 取樣操作需在冰上進行,避免樣本的凍融,全程操作器械預先進行 RNase free 處理,採集後將樣本分割成100 mg左右的小塊,並立即放入預冷 RNAlater 按照使用手冊逐步冷凍,保存管使用 parafilm 封口,-80℃ 保存。
- 若採樣組織 RNA 表現量不高,適情況增加收取樣本量,確保萃取核酸足夠 RNAseq 使用。(例:脂肪、骨頭等組織。)
- 細胞收取前,可先用 RNase free 預冷 PBS 輕微沖洗細胞表層,移除懸浮死細胞和多餘培養液,並使用預冷 TRI reagent 沖洗方式收取細胞,並使用 pipetting 方式確實均質溶液後 -80℃ 保存。
- 植物樣本採樣時,須排除可能的環境微生物/昆蟲汙染,若水生/潮濕環境生長植物,必要時需要進行抗生素培養。
RNAseq雲平台-線上分析工具
RNAseq雲平台功能介紹:
▶ 支援多物種分析
目前可支援 32 種
▶ 把圖變成你喜歡的顏色與形狀 直接在平台上調整製圖大小、標題、顏色、字體
提供多種下載格式,製作報告好簡單
▶ 任你自由調整的篩選門檻與參數 線上調整分組及樣品名稱 調整p-value、Fold Change等等篩選門檻 ▶ 一種分析,多種呈現方式 任意選擇適合你的呈現方式 (長條圖、富集點圖、網路圖)
▶ 最豐富的分析項目
差異基因分析、基因集富集分析(GO、KEGG、DO、GSEA)、蛋白交互作用分析
各項資料庫版本持續更新
▶ 讓分析結果最大化,資料庫超連結有夠方便 點擊ID名稱立即連動資料庫,各項資訊一目了然
▶ 客製化熱圖自己動手做 上傳篩選基因清單,自訂標準化方法、聚類方向即輕鬆完成
觀看RNAseq雲平台解說文章
觀看RNAseq雲平台直播影片
圖爾思雲平台入口
▶ 支援多物種分析
目前可支援 32 種
▶ 把圖變成你喜歡的顏色與形狀 直接在平台上調整製圖大小、標題、顏色、字體
提供多種下載格式,製作報告好簡單
▶ 任你自由調整的篩選門檻與參數 線上調整分組及樣品名稱 調整p-value、Fold Change等等篩選門檻 ▶ 一種分析,多種呈現方式 任意選擇適合你的呈現方式 (長條圖、富集點圖、網路圖)
▶ 最豐富的分析項目
差異基因分析、基因集富集分析(GO、KEGG、DO、GSEA)、蛋白交互作用分析
各項資料庫版本持續更新
▶ 讓分析結果最大化,資料庫超連結有夠方便 點擊ID名稱立即連動資料庫,各項資訊一目了然
▶ 客製化熱圖自己動手做 上傳篩選基因清單,自訂標準化方法、聚類方向即輕鬆完成
觀看RNAseq雲平台解說文章
觀看RNAseq雲平台直播影片
圖爾思雲平台入口