WGS vs. WES
全基因體定序 (whole genome sequencing, WGS) 與全外顯子定序 (whole exome sequencing, WES) 該選哪種呢?
(圖片來源http://www.genomesop.com/somatic-mutations/)
相信這個問題一定很多研究人員都曾想過,其實標準答案就是--------------- 如果有足夠經費的話,當然選WGS啊!!! (那大家看到這邊就可以關視窗了) 不,請等一下,經費當然永遠嫌不足!
先讓我們來深入了解一下 WGS 與 WES 兩者之間的差異~ 人類基因體的組成內容 人類基因體大小約為3 x 109 bp (30億),Protein-coding sequences 大約占整個基因體的1.5%,而剩下的區域大致上可分為:
- non-coding RNA genes
- regulatory DNA sequences
- introns
- LINEs (long interspersed nuclear elements)
- SINEs (short interspersed nuclear elements)
- sequences for which as yet no function has been determined
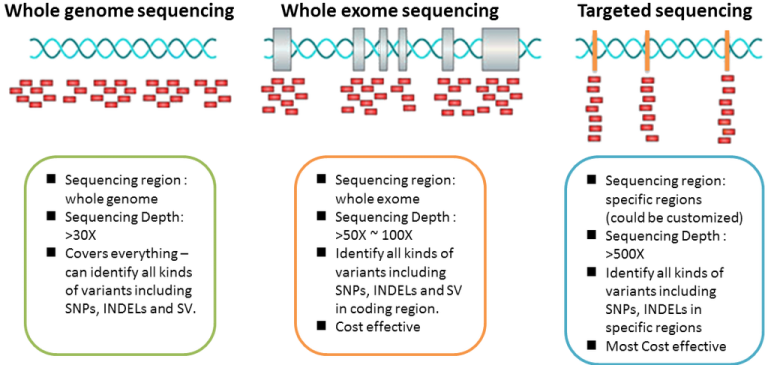
DNA定序的種類 而目前利用NGS (next generation sequencing) 技術應用在DNA定序上的種類,大致可以分成三類:
- Whole genome sequencing是將整個人類基因體都拿來定序,但是由於定序技術上的限制,有些區域還是比較難被定序到,例如: 高GC的區域(high GC content)、大片段重複的區域(large repeat regions )、 染色體的中節(centromeres)和端粒(telomeres),所以實際大約是涵蓋整個基因體的95-98%區域。
- Whole exome sequencing是針對蛋白質編碼的區域(protein coding sequences)進行定序。
- Target sequencing是針對特定區域的範圍進行定序,例如常見的癌症基因定序(cancer panel),就是針對與癌症相關的基因進行定序。
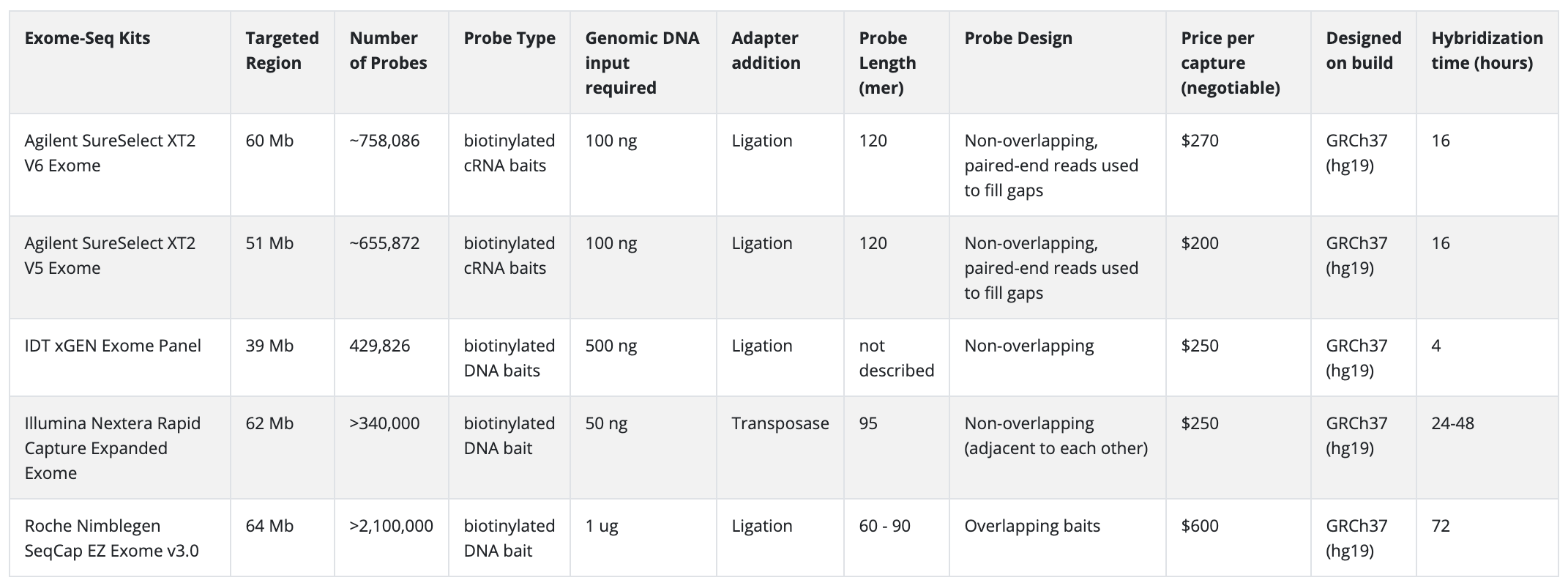
(表一)列舉了一些目前常見的exome enrichment kit,並且比較了其覆蓋的區域大小和相關資訊,由資料可知目前市面上常見的幾種全外顯子設計涵蓋的區域約為 39-64Mb 左右,需要的 DNA 量與實驗上需要的時間都不相同。
表一:目前常見幾種 exome enrichment kit 間的差異
表格來源: https://genohub.com/exome-sequencing-library-preparation/
WGS與WES在定序深度(coverage)的差異
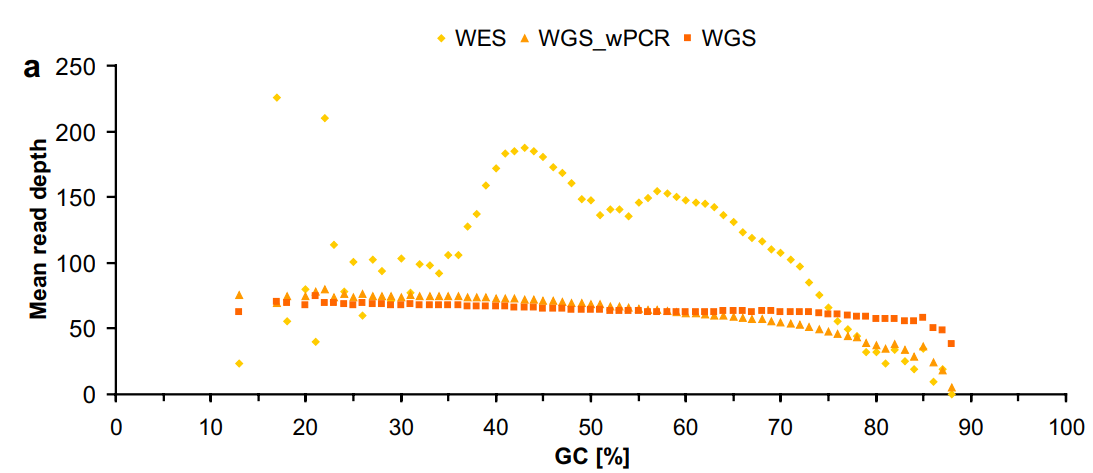
(表二)是illumina官方網站上提供的 WGS 和 WES 建議的定序深度,分別為 30x-50x 和 100x。 為什麼會有這種差別呢? 為什麼除了定序的區域不同,建議的定序深度也要不一樣呢?這就要提到前面所說的,WES在上機定序之前必須先經過捕獲(Capture)和擴增(Enrichment)的步驟,就是這個步驟會有效率的問題,有些exon區域捕獲的效率很高,有些 exon 區域捕獲效率很低 (圖二和圖三) [1],加上人類主要的 exon 長度約 200bp [2],所以定序長度如果大於 200bp 的話,那多餘的部分其實就是浪費掉了,所以經過大家統計之後發現,WES 實際產出的定序量只有 65%-75% 會是落在 exome 區域,所以只好多產出一些定序量來提高那些捕獲率較低的區域以及浪費掉的定序片段。
表二: Illumina官方對不同定序種類 coverage 的建議 
圖二:研究中比較了 WES、WGS_wPCR(WGS需要PCR步驟)和 WGS(不需要 PCR 步驟)對於 GC 含量差異與定序深度之間的關係。可以發現 WGS(不需要 PCR 步驟)也就是方形深橘色軌跡在不同 GC% 其平均定序深度都是相當地一致,而 WES 在不同 GC% 的平均定序深度變動很大,這就會造成定序深度不一致的問題。
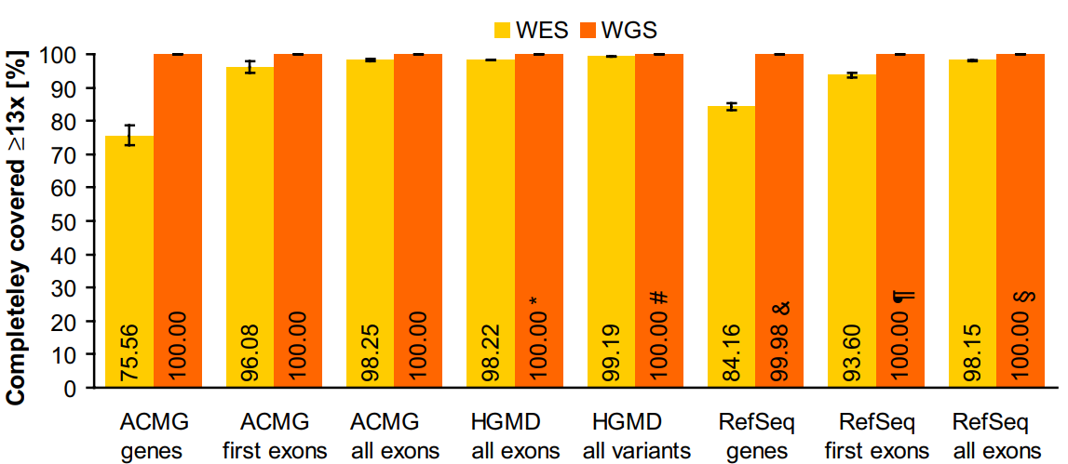
圖三:WGS 的定序深度並不會在基因體中有明顯差異,而 WES 在第一個exon的定序深度明顯比 WGS 低
生物資訊分析上的差異 目前在分析 single nucleotide variation (SNV) 與 insertion and deletion (Indel) 上,資料來源是WGS或是WES用的分析工具是相同的。不過在分析大片段的變異,例如:拷貝數變異 (copy number variation, CNV)以及結構變異(structural variation, SV),有些專門為 WES 分析設計的工具,例如 exomeCNV。不過大部分的工具都可以適用 WGS 和 WES 資料,例如:GATK CNV,差別是在分析 WES 資料時,需要給定 exome 的區間資料。不過根據本篇之前提到的,WGS 的優勢在於對於基因體覆蓋度高並且一致性也較高,分析出來的結果當然比較全面並且可信度較高囉。
在臨床檢測上的應用
(1) 目前臨床檢測上常見的方式是,第一階段先用 gene panel 定序的方式尋找突變,如果找不到,接下來第二階段就利用 WES 或是 WGS 來尋找與疾病相關突變 [1]。會這麼做是因為 gene panel 價格便宜、分析快,而且是根據前人研究挑選出與疾病有關的基因,容易找到目標突變位置。
(2) 與 WES 相比,WGS 的優勢在於可以找到可信度較高的「拷貝數變異」和「結構變異」,而這些很可能就是跟疾病有關的變異(因為容易造成基因異常表現),加上最近這種 PCR-free WGS 技術,可以更完整地覆蓋整個基因體,使得整個基因體覆蓋度一致的情況下,WGS更適合於偵測大片段的變異。
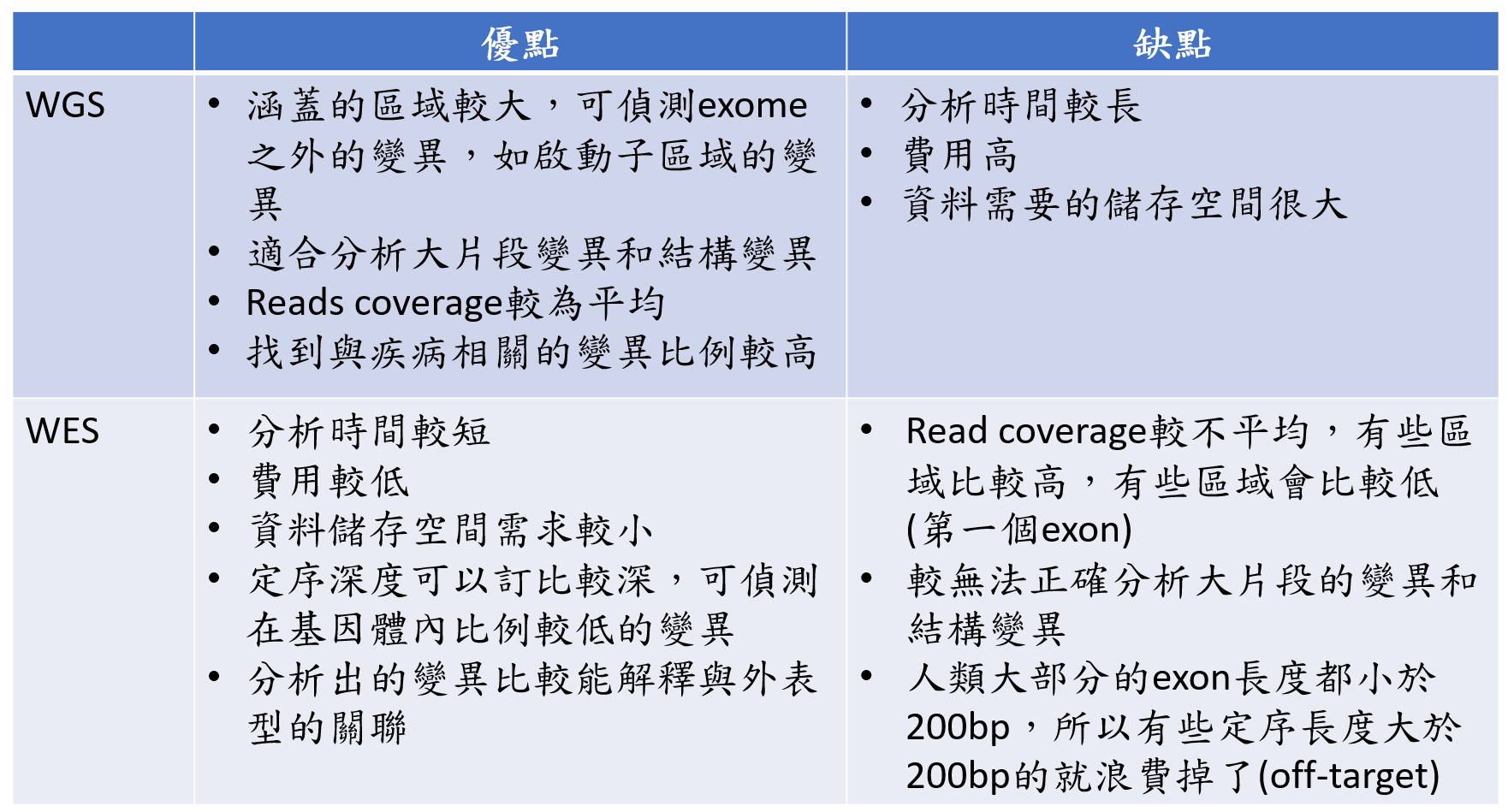
所以整體而言,在不考慮費用的情況之下,WGS是優於WES的,可以更全面地分析疾病的可能原因 (表三) [6]。
表三:WGS與WES疾病分析比較
最後,小編以表格的方式幫大家比較這兩種定序的差異~
圖爾思生物科技 / 微生物體研究中心