13
2016.06
Metagenome定序中的unmapping reads - 把垃圾變黃金
眾所皆知,青黴素在二戰時期拯救了無數人的生命,也讓發現者弗萊明(Alexander Fleming,1881-1955)獲得了諾貝爾醫學獎。若當年,弗萊明把無意間混入青黴素的培養皿當做垃圾丟掉,那麼人類將可能失去這個醫療上的偉大發現!
在高通量定序領域中,由於技術侷限,宏基因組(Metagenome)定序中的unmapping reads往往被當成『垃圾資料』而忽略掉。然而,我們從三篇Nature文章中找到了如何將宏基因組中的垃圾資料變成黃金的秘訣~
混合組裝 (Mix assembly)
三篇分別發表於Nature[1,2]與Nature Communication[3]的文章,全部應用了基於unmapping reads的混合組裝。那麼混合組裝究竟有什麼優勢呢?
優勢1. 充分利用定序資料,增加reads的利用率
優勢2. 挖掘低豐度物種和基因資訊
我們來結合圖2舉例說明一下:
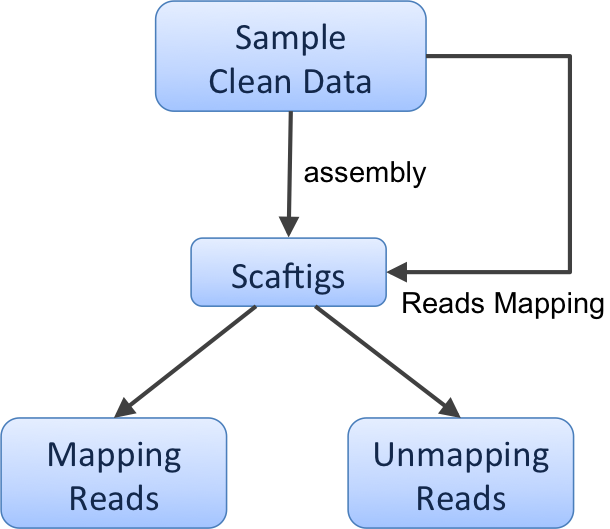
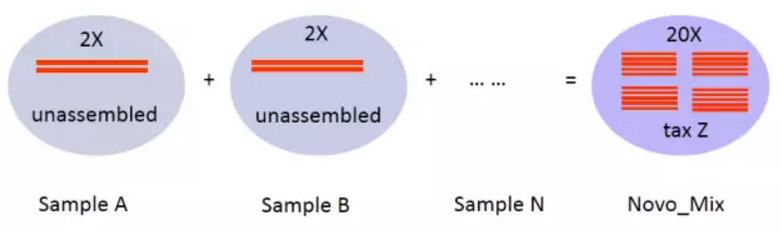
若有N (N=10) 個定序樣品A...N,經過QC後,各樣品單獨組裝成scaftigs。
假設每個樣品中低豐度物種 z 的定序深度都是2x,這意味著它們很難被組裝,因此物種 z 的定序reads成為了unmapping reads。我們將這10個樣品的unmapping reads合併起來,加入混合組裝流程,物種 z 的定序深度則可達到20x,這樣就提高了這類低豐度物種序列的可組裝性,並增大了發現低豐度物種的可能性。
三篇分別發表於Nature[1,2]與Nature Communication[3]的文章,全部應用了基於unmapping reads的混合組裝。那麼混合組裝究竟有什麼優勢呢?
優勢1. 充分利用定序資料,增加reads的利用率
優勢2. 挖掘低豐度物種和基因資訊
我們來結合圖2舉例說明一下:
若有N (N=10) 個定序樣品A...N,經過QC後,各樣品單獨組裝成scaftigs。
假設每個樣品中低豐度物種 z 的定序深度都是2x,這意味著它們很難被組裝,因此物種 z 的定序reads成為了unmapping reads。我們將這10個樣品的unmapping reads合併起來,加入混合組裝流程,物種 z 的定序深度則可達到20x,這樣就提高了這類低豐度物種序列的可組裝性,並增大了發現低豐度物種的可能性。
因此,小編認為:如何有效的利用這些unmapping reads在Metagenome中顯得相當重要,相信往後發表的研究中,都會使用此混合組裝技術來將過去的垃圾變成黃金的!!
1. Qin J, Li R, Raes J, et al . A human gut microbial gene catalogue established by metagenomic sequencing. Nature . 2010, 464: 59-65.
2. Karlsson FH, Tremaroli V, Nookaew I,et al . Gut metagenome in European women with normal, impaired and diabetic glucose control. Nature .2013, 498: 99–103.
3. Karlsson FK, Fak F, Nookaew I, et al . Symptomatic atherosclerosis is associated with an altered gut metagenome. Nat Commun . 2012, 3:1245.