13
2020.02
普氏分析 (Procrustes analysis)
原創文章 引用請註明出處
多體學(Multiomics)是將多筆體學資料,像是轉錄體(transcriptome)、 蛋白質體(proteome)、微生物體(microbiome)、代謝體(metabolome)... 等整合研究,藉以推測潛在的因果關係(文章回顧:多體學新思維)。然而,多資料整合也有可能會遇上兩資料集相互無法解釋的情況。假設我們手中有代謝體資料跟微生物體資料,要如何得知「微生物群落結構預測代謝變化的可行性呢」?這時候普氏分析(Procrustes Analysis)就很好用了。
多體學(Multiomics)是將多筆體學資料,像是轉錄體(transcriptome)、 蛋白質體(proteome)、微生物體(microbiome)、代謝體(metabolome)... 等整合研究,藉以推測潛在的因果關係(文章回顧:多體學新思維)。然而,多資料整合也有可能會遇上兩資料集相互無法解釋的情況。假設我們手中有代謝體資料跟微生物體資料,要如何得知「微生物群落結構預測代謝變化的可行性呢」?這時候普氏分析(Procrustes Analysis)就很好用了。
小故事介紹
普洛克路斯忒斯(Procrustes)是希臘神話中的一名強盜,他開了一間旅店,宣稱店裡的床鋪吻合所有人的身高,但作法其實是將身高長者截短、身高短者強行拉長。簡而言之,這個神話裡的強盜就是在「削足適履」,將兩個不一樣的屬性安裝到一個標準上 [1]。數學統計中的普氏分析就有點類似這種概念,我們可以這麼想像:
數學模型
那麼計算上具體做了什麼呢?
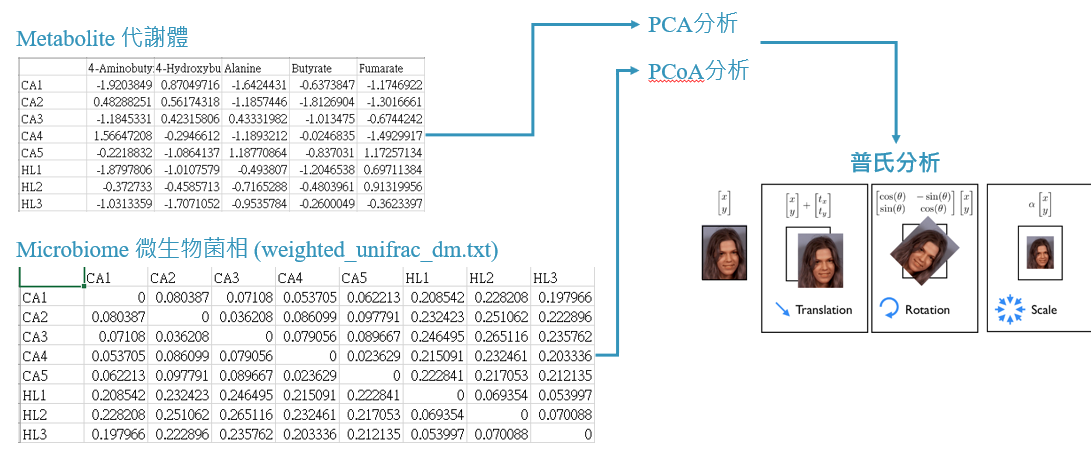
普氏分析是一種形狀分佈分析方法[3, 4],利用PCA、PCoA、NMDS等降維排序對多體學資料做視覺化呈現,透過平移、旋轉、縮放將兩個資料集的主成分疊加到低維空間(Procrustes Superimposition),以便快速檢視多體學資料的一致性。
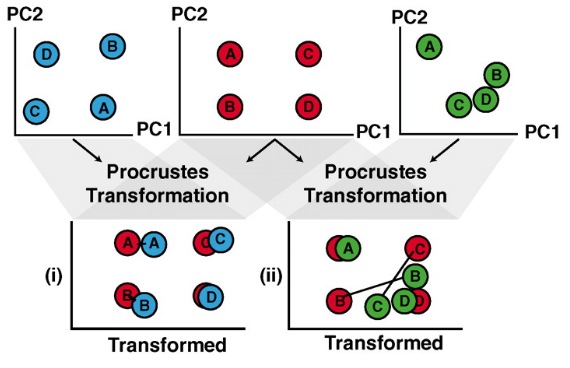
如下圖[5],這邊有三個資料集,以藍/ 紅/ 綠示意。 (i) 來自紅色與藍色的點雖然原始位置分配不太一樣,但經過Procrustes Transformation,可斷定來自藍色與紅色點的資料有較佳的吻合度,彼此可以互相呼應;相反地,(ii) 紅色與綠色的點經轉換後,某些同樣本的點間距離仍是很遠,相較於(i),紅/ 綠資料可能較無法互相解釋。
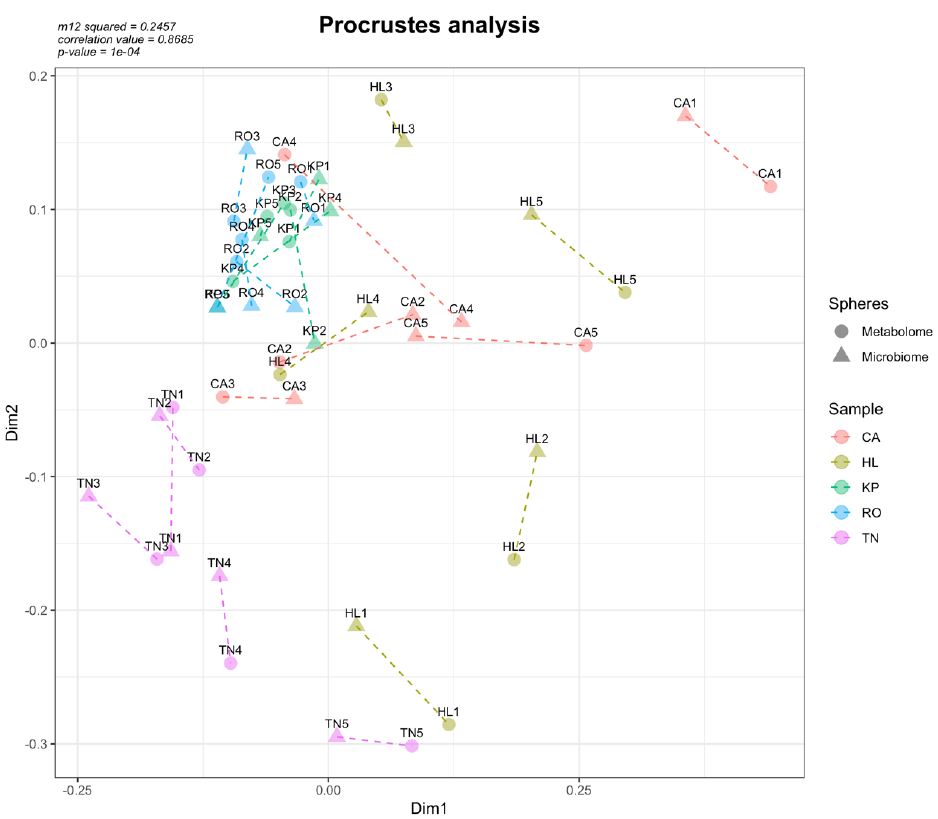
承前面所述之資料,假設我們手邊有來自五組樣本(以顏色區分,每組又各有5個樣本)各自的微生物體資料與代謝體資料(以形狀區分),利用普氏分析(示意圖如下),我們將代謝體資料投射到微生物體資料((un)Weighted UniFrac Distance)上。 封面照片即為分析結果。圖中,每個相連的點為同一個樣本,來自代謝體/ 微生物體。連線長度表示菌群和代謝體資料的差異,連線越長差異越大。m12 sqaured 是經過疊加後在兩個排序空間中對應點間的距離平方和。Correlation value 是 Procrustes 分析基於squared m12值計算得到的相關性係數。p-value 則是經 9,999 次置換檢驗(permutation)所得到的相關性顯著性數值。

應用性
目前普氏分析有哪些應用呢?
(1) 將代謝體的分佈透過旋轉縮放映射到菌相組成的分佈上,便可同時觀察不同樣品菌相結構的差異和代謝物組成的差異[7-9]
(2) 不同體學間相似性比較,如微生物、KEGG、CAZy等[5]
(3) 對同一份資料做兩種不同的排序分析(例如PCA與PCoA)或不同的研究目的或目標(例如功能基因)[10, 11],以普氏分析評估兩次分析結果的相關性。
例如:比較不同腸段微生物總體基因體與代謝體資料,發現兩種體學資料的相關性在盲腸高於乙狀結腸[12]
看完普氏分析介紹是不是覺得多體學分析很有趣呢?來看看其他多體學相關文章吧:
目前普氏分析有哪些應用呢?
(1) 將代謝體的分佈透過旋轉縮放映射到菌相組成的分佈上,便可同時觀察不同樣品菌相結構的差異和代謝物組成的差異[7-9]
(2) 不同體學間相似性比較,如微生物、KEGG、CAZy等[5]
(3) 對同一份資料做兩種不同的排序分析(例如PCA與PCoA)或不同的研究目的或目標(例如功能基因)[10, 11],以普氏分析評估兩次分析結果的相關性。
例如:比較不同腸段微生物總體基因體與代謝體資料,發現兩種體學資料的相關性在盲腸高於乙狀結腸[12]
看完普氏分析介紹是不是覺得多體學分析很有趣呢?來看看其他多體學相關文章吧:
參考文獻
[1] https://ccjou.wordpress.com/2013/11/21/%E6%AD%A3%E4%BA%A4-procrustes-%E5%95%8F%E9%A1%8C/
[2] http://www.mythweb.com/today/media/procrustes07.gif
[3] Cattell, J.R.H.R.B., The procrustes program: Producing direct rotation to test a hypothesized factor structure. Behavioral Science, 1962. 7(2): p. 258-262.
[4] Jackson, P.R.P.-N.A., How well do multivariate data sets match? The advantages of a Procrustean superimposition approach over the Mantel test. Oecologia, 2001. 129(2): p.169-178.
[5] Muegge, B.D., et al., Diet drives convergence in gut microbiome functions across mammalian phylogeny and within humans. Science, 2011. 332(6032): p. 970-4.
[6] https://blog.csdn.net/jinshengtao/article/details/43376049
[7] Quinn, R.A., et al., From Sample to Multi-Omics Conclusions in under 48 Hours. mSystems, 2016. 1(2).
[8] Ayeni, F.A., et al., Infant and Adult Gut Microbiome and Metabolome in Rural Bassa and Urban Settlers from Nigeria. Cell Rep, 2018. 23(10): p. 3056-3067.
[9] McCall, L.I., et al., Experimental Chagas disease-induced perturbations of the fecal microbiome and metabolome. PLoS Negl Trop Dis, 2018. 12(3): p. e0006344.
[10] Dhakan, D.B., et al., The unique composition of Indian gut microbiome, gene catalogue, and associated fecal metabolome deciphered using multi-omics approaches. Gigascience, 2019. 8(3).
[11] Manor, O. and E. Borenstein, Revised computational metagenomic processing uncovers hidden and biologically meaningful functional variation in the human microbiome. Microbiome, 2017. 5(1): p. 19.
[12] McHardy, I.H., et al., Integrative analysis of the microbiome and metabolome of the human intestinal mucosal surface reveals exquisite inter-relationships. Microbiome, 2013. 1(1): p.17.
圖爾思生物科技 / 微生物體研究中心
謝馥媺 文案