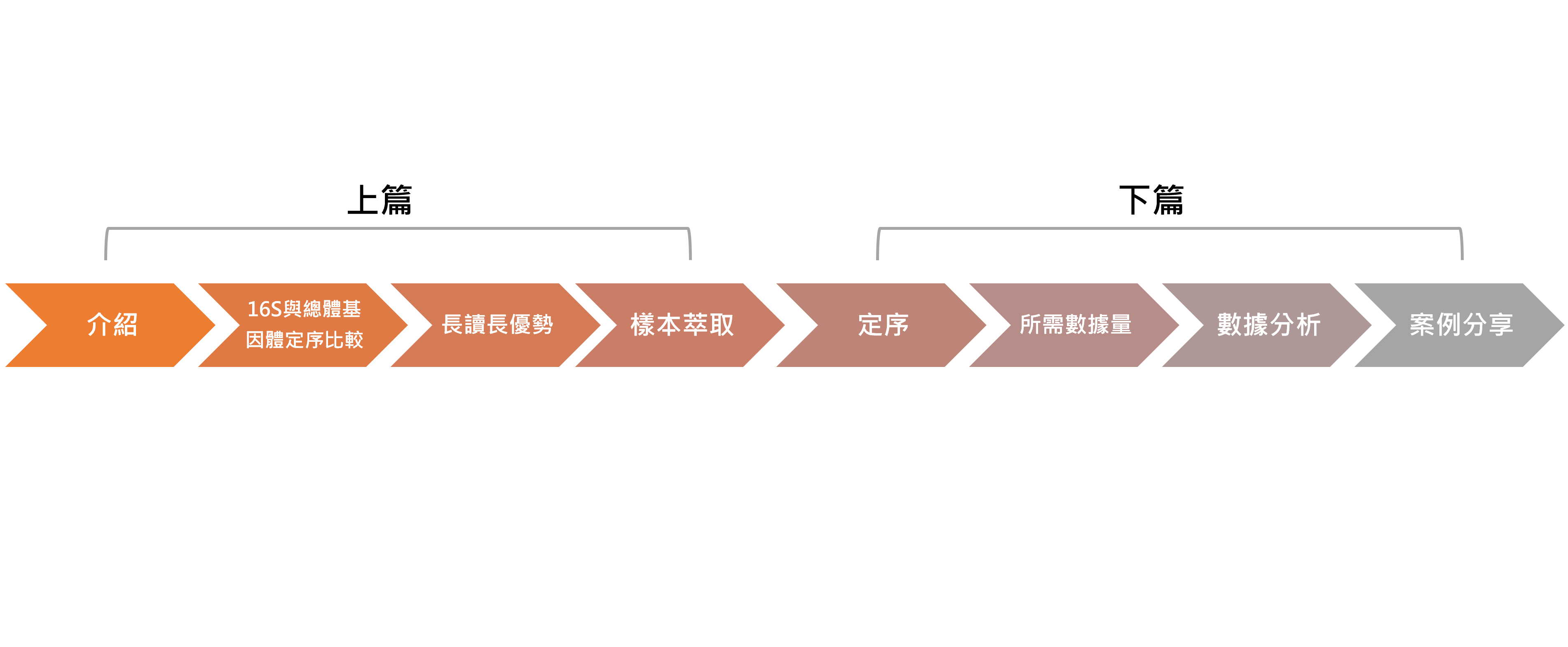
08
2021.12
你。不只是你。總體基因體學揭開人體內宇宙 (上)
原創文章 引用請註明出處
『我不怕,就算殺了一個我,還有千千萬萬個我。』~出自「功夫」的…理髮師。 今天小編就從科學上來跟你說其實這句話一點也不誇飾,甚至還含蓄了些。
你。不只是你。
如果說人體就是一個宇宙的話,這個宇宙的成員是細胞和各種各樣的細菌,數量甚至遠遠大於銀河系的恆星。即使是在你很乾淨也很健康的狀態時,你的皮膚上每時每刻也有一萬億個細菌在覓食。算算比例,人體大約由一億億個細胞組成,還有10億億個細菌寄生。聽起來有沒有比漫威宇宙還要廣闊複雜 !? (原諒小編最近看了永恆族又被洗腦了 XDDD)
沒錯,我們只有 10% 是人類 [1]。
這些體內微生物的組成對於我們的健康具有指標上的意義。人與人之間基因體的差異僅有 0.1%,而互相的微生物居民組成卻有 80~90% 的差異 (Fig. 1)。其組成也會隨著人生階段、飲食、健康狀態等而改變 (Fig. 2),近年更發現其與多種疾病具有高度相關性,因此,監控或調控人體微生物組成也成為了控制疾病風險的一種新興手段。
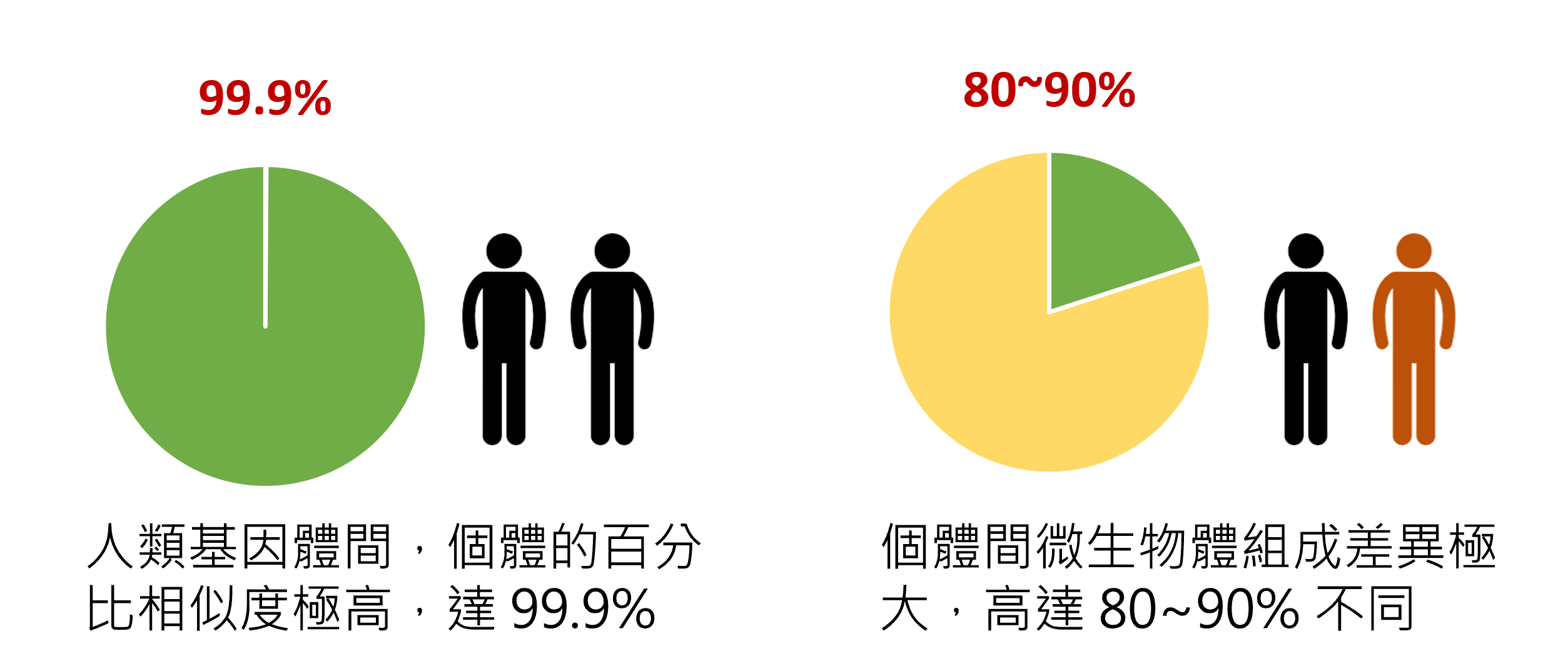
(Fig. 1)
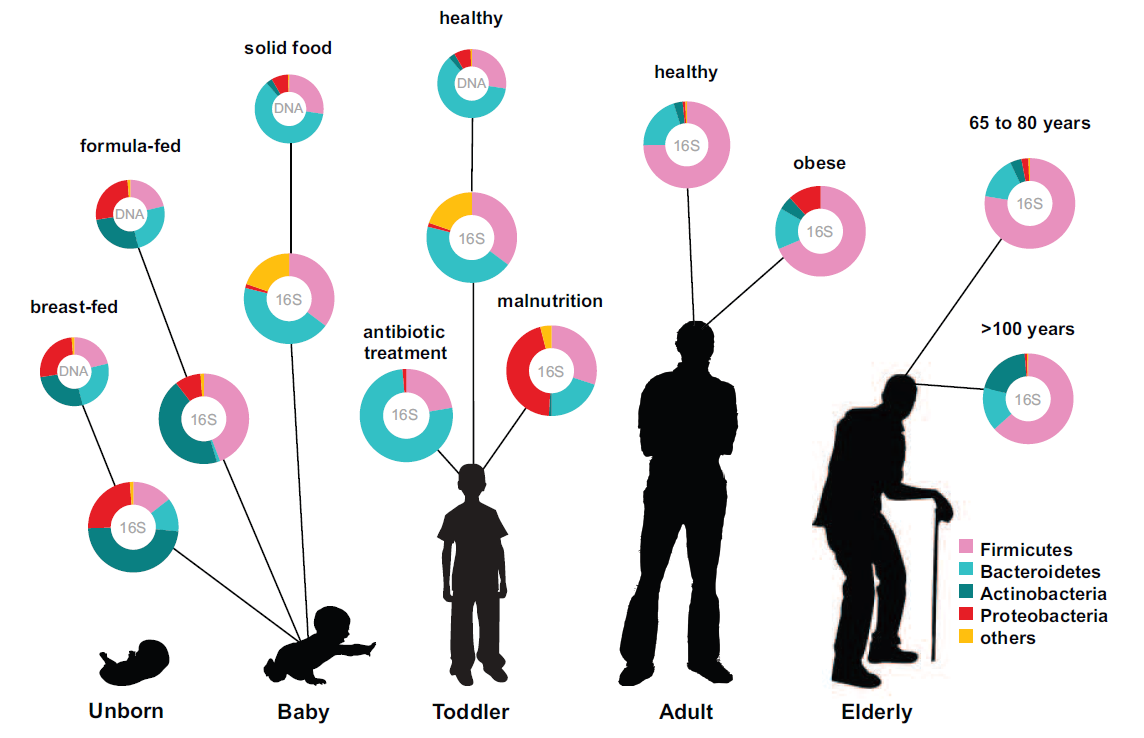
(Fig. 2) 圖片來源 [2]
而在科學上要怎麼挖掘出另外 90% 的宇宙組成呢? 定序是一個很好的方法,依據目的性有兩個方法可以選擇,一是"16S rRNA gene定序",其可以藉由定序細菌 16S 片段知曉細菌群落的組成,但此方法僅能解析至菌相組成且限於細菌,詳細介紹請左轉先前好文” 全長16S rRNA定序-從屬進階到種的新高度”
而另一種方法可以同時看到細菌、古細菌、病毒等微生物,且不僅能解析菌落組成還能夠進行抗藥性等功能性分析。這種從單個生物體中獲得的多種生物體的基因體分析樣本,通常被稱為“總體基因體學”,允許很好的深入了解微生物群落的遺傳組成。傳統上,微生物研究是通過使用人工培養單個物種或菌株; 然而,據估計只有不到 2% 的細菌可在實驗室培養,而定序可以跳過培養的障礙直接對微生物遺傳物質進行解密。
各自的優缺比較可參考下表:

在結果組裝方面,總體基因體定序難度不是一般單菌組裝可以比擬,單菌組裝就像是完成一份拼圖; 而總基因體組裝就是上百甚至上千份拼圖混在一起拼裝...有沒有光想就覺得放棄比較輕鬆 (¬‿¬) 但是! 如果這些拼圖都是幼兒拼圖等級的大塊拼圖呢? 有沒有突然想衝動喊出: 老闆,這樣的拼圖給我來一打!!! 長讀長定序的優勢就在於將高難度拼圖轉變成幼兒拼圖。試想,一樣面積的拼圖,今天每一塊拼圖的大小為 50 bp,就需要拚滿 92,000 塊拼圖才能完成; 但若每一塊拼圖的大小為 500,000 bp,僅只需要 9 塊拼圖就可以完美組裝 (Fig. 3)。
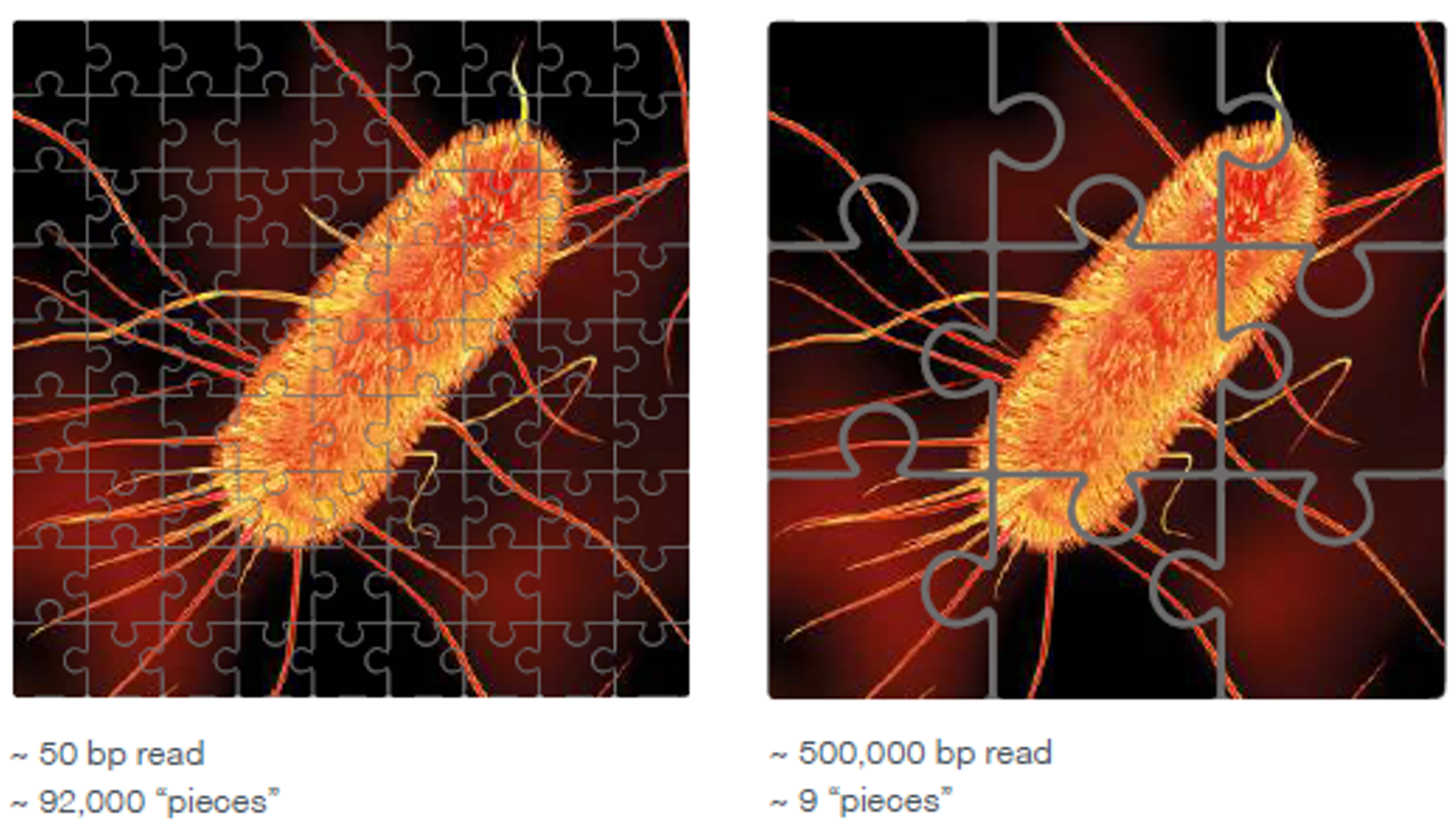
(Fig. 3)
當然基因體的組裝比拼圖複雜的多,除了組裝的完整度,尚需要考慮技術選擇上有沒有可能造成系統偏差。因此,總體基因體的研究上,可以選擇使用 Nanopore 長讀長定序技術,其改善了短讀長定序所提供的傳統基因散彈槍法總體基因體分析,優點整理如下:
1. 容易且無偏好性組裝,包含完整基因體與質體
2. 區分密切相關的種屬
3. 解析具挑戰性之重複區域和結構變異
4. 解析抗生素耐藥性 (AMR) 基因及毒力因子等
5. 具成本效益
6. 即時數據分析,快速提供樣本結果
看完上列介紹,如果發現總體基因體才是你的需求、能解答你研究的目的,那麼我們第一件事就是要獲取微生物群落的高分子量 DNA (High Molecular Weight DNA, HMW DNA) 。(不是你目前的需求也可以繼續看下去~俗話說: 人如果不上進,跟鹹魚又有什麼分別?---修改自<少林足球>)
由於原始片段越長進行長讀長定序才有其意義,所以長讀長定序在樣品完整度上要求較短讀長嚴格許多。但是從不同的樣本中萃取完整 DNA 有其困難度,因此 Nanopore 針對不同樣本收集了建議的 HMW DNA 萃取方法於Nanopore community (Fig. 4),萃取 protocol 包含土壤、糞便、人類、多種動植物等樣本類型。此外,還提供了不同類型樣本儲存、DNA/RNA 處理、建庫準備等主題,以確保研究者可以獲取最高品質之樣本以便進行長讀長定序。
(Fig. 4)Nanopore 提供之 HMW DNA 萃取方法
以總體基因體定序常見之糞便樣本為例,要從人類糞便中萃取高度完整性、足夠總量、純度的 DNA 用於總體基因體定序仍然是一項長期研究障礙。
Nanopore 以大鼠糞便為例介紹了四種萃取 DNA 的方法 [3]。當目標為宿主(例如大鼠)時,推薦使用 QIAGEN DNeasy Blood and Tissue Kit,當目標為微生物時推薦使用 QIAamp PowerFecal DNA Kit。而使用 QIAGEN Genomic-tip 20/G 和 QIAGEN MagAttract HMW DNA Kit 則可同時觀察到比例相近的宿主和微生物 (Fig. 5) 。比較結果顯示,Genomic-tip 方法產生較長之 read length (Fig. 6) ,而 MagAttract HMW DNA Kit 萃取流程耗時較短。
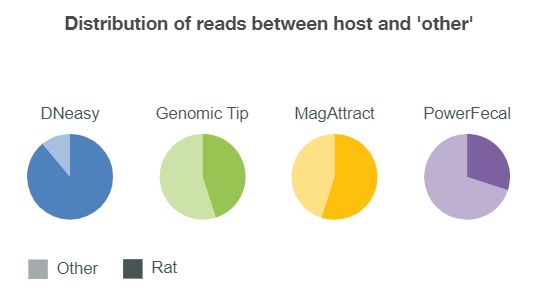
(Fig. 5)Nanopore 推薦核酸萃取套組之萃取組成結果
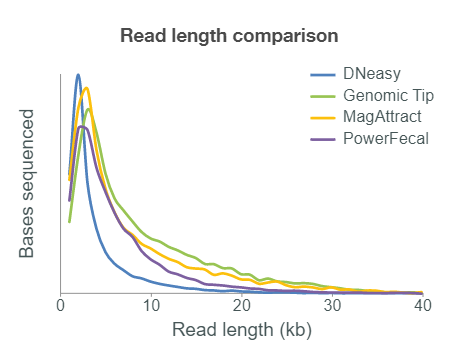
(Fig. 6)Nanopore 推薦核酸萃取套組之定序讀長結果分布

(Fig. 7) Nanopore 推薦核酸萃取套組之比較
另外補充,除了商業化的核酸萃取套組,欲萃取高品質人類糞便樣品的研究者也可參照 2020 發表於 nature protocols 之文獻 [4],研究團隊提出之萃取 protocol 極致詳盡,包含多個酵素裂解及片段篩選步驟。可由總量 300-500mg 的樣品中萃取 1-2 µg 的 HMW DNA,片段範圍坐落於 15-50 kb,於 MinION R9.4 上可生成約 6-30 Gbp 的數據。除了詳列所需試劑與萃取步驟,也貼心列出可能遇到的狀況與解決方法,總萃取時間可在 8 小時完成。(Fig. 8)
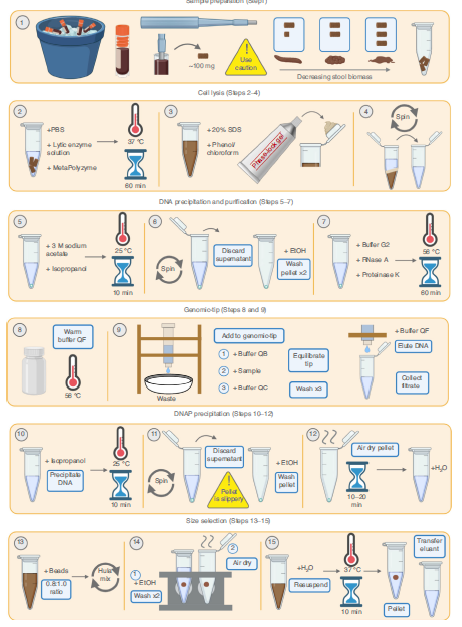
(Fig. 8)
此外,很重要的一點是在進行總體基因體定序前需要注意宿主 DNA 的汙染。在定序前移除宿主 DNA 汙染可避免浪費極大部分的定序數據被宿主 DNA 佔據。 舉例來說,人類下呼吸道感染(LRIs) 的樣本帶有大量的宿主細胞 (宿主 : 病原體 = 100,000 : 1) 對於使用總體基因體定序進行 LRIs 患者致病菌鑑定造成執行上的極大障礙。其解決方法可參考 2019 年發表於 nature biotechnology 之研究 [5],其研究團隊使用皂素為基底的方法可從臨床呼吸道樣本中去除高達 99.99% 的宿主核酸,搭配 Nanopore 定序及調整實驗流程,從樣本至結果可於 6 小時內準確識別病原體和抗生素抗性基因。(詳細案例分享請參照下篇)
本篇文章希望能以深入淺出的描述,讓大家體會到使用長讀長 Nanopore 定序進行總體基因體研究的威力,至今已經有許多研究團隊開發出使用 Nanopore 快速檢測臨床上致病原的方法,(下) 篇再帶大家導覽相關的案例分享,以及不同目的需求所需的定序數據量與 Nanopore 提供的分析流程。 那麼,元旦後 (下) 集見~
傳送門: 你。不只是你。總體基因體學揭開人體內宇宙 (下)

參考資料
1. 我們只有10%是人類:認識主宰你健康與快樂的90%細菌,https://www.books.com.tw/products/0010710936
2. Ottman N, Smidt H, de Vos WM, Belzer C. The function of our microbiota: who is out there and what do they do? FrNanopore Cell Infect Microbiol. 2012 Aug 9;2:104. doi: 10.3389/fcimb.2012.00104. PMID: 22919693; PMCID: PMC3417542.
3. Stool pellets extraction method comparison, https://community.Nanoporetech.com/extraction_methods/stool_pellets
4. Maghini, D.G., Moss, E.L., Vance, S.E. et al. Improved high-molecular-weight DNA extraction, Nanopore sequencing and metagenomic assembly from the human gut microbiome. Nat Protoc 16, 458–471 (2021). https://doi.org/10.1038/s41596-020-00424-x
5. Charalampous, T., Kay, G.L., Richardson, H. et al. Nanopore metagenomics enables rapid clinical diagnosis of bacterial lower respiratory infection. Nat Biotechnol 37, 783–792 (2019). https://doi.org/10.1038/s41587-019-0156-5

『我不怕,就算殺了一個我,還有千千萬萬個我。』~出自「功夫」的…理髮師。 今天小編就從科學上來跟你說其實這句話一點也不誇飾,甚至還含蓄了些。
你。不只是你。
如果說人體就是一個宇宙的話,這個宇宙的成員是細胞和各種各樣的細菌,數量甚至遠遠大於銀河系的恆星。即使是在你很乾淨也很健康的狀態時,你的皮膚上每時每刻也有一萬億個細菌在覓食。算算比例,人體大約由一億億個細胞組成,還有10億億個細菌寄生。聽起來有沒有比漫威宇宙還要廣闊複雜 !? (原諒小編最近看了永恆族又被洗腦了 XDDD)
沒錯,我們只有 10% 是人類 [1]。
這些體內微生物的組成對於我們的健康具有指標上的意義。人與人之間基因體的差異僅有 0.1%,而互相的微生物居民組成卻有 80~90% 的差異 (Fig. 1)。其組成也會隨著人生階段、飲食、健康狀態等而改變 (Fig. 2),近年更發現其與多種疾病具有高度相關性,因此,監控或調控人體微生物組成也成為了控制疾病風險的一種新興手段。

(Fig. 1)

(Fig. 2) 圖片來源 [2]
而在科學上要怎麼挖掘出另外 90% 的宇宙組成呢? 定序是一個很好的方法,依據目的性有兩個方法可以選擇,一是"16S rRNA gene定序",其可以藉由定序細菌 16S 片段知曉細菌群落的組成,但此方法僅能解析至菌相組成且限於細菌,詳細介紹請左轉先前好文” 全長16S rRNA定序-從屬進階到種的新高度”
而另一種方法可以同時看到細菌、古細菌、病毒等微生物,且不僅能解析菌落組成還能夠進行抗藥性等功能性分析。這種從單個生物體中獲得的多種生物體的基因體分析樣本,通常被稱為“總體基因體學”,允許很好的深入了解微生物群落的遺傳組成。傳統上,微生物研究是通過使用人工培養單個物種或菌株; 然而,據估計只有不到 2% 的細菌可在實驗室培養,而定序可以跳過培養的障礙直接對微生物遺傳物質進行解密。
各自的優缺比較可參考下表:

在結果組裝方面,總體基因體定序難度不是一般單菌組裝可以比擬,單菌組裝就像是完成一份拼圖; 而總基因體組裝就是上百甚至上千份拼圖混在一起拼裝...有沒有光想就覺得放棄比較輕鬆 (¬‿¬) 但是! 如果這些拼圖都是幼兒拼圖等級的大塊拼圖呢? 有沒有突然想衝動喊出: 老闆,這樣的拼圖給我來一打!!! 長讀長定序的優勢就在於將高難度拼圖轉變成幼兒拼圖。試想,一樣面積的拼圖,今天每一塊拼圖的大小為 50 bp,就需要拚滿 92,000 塊拼圖才能完成; 但若每一塊拼圖的大小為 500,000 bp,僅只需要 9 塊拼圖就可以完美組裝 (Fig. 3)。

(Fig. 3)
當然基因體的組裝比拼圖複雜的多,除了組裝的完整度,尚需要考慮技術選擇上有沒有可能造成系統偏差。因此,總體基因體的研究上,可以選擇使用 Nanopore 長讀長定序技術,其改善了短讀長定序所提供的傳統基因散彈槍法總體基因體分析,優點整理如下:
1. 容易且無偏好性組裝,包含完整基因體與質體
2. 區分密切相關的種屬
3. 解析具挑戰性之重複區域和結構變異
4. 解析抗生素耐藥性 (AMR) 基因及毒力因子等
5. 具成本效益
6. 即時數據分析,快速提供樣本結果
看完上列介紹,如果發現總體基因體才是你的需求、能解答你研究的目的,那麼我們第一件事就是要獲取微生物群落的高分子量 DNA (High Molecular Weight DNA, HMW DNA) 。(
由於原始片段越長進行長讀長定序才有其意義,所以長讀長定序在樣品完整度上要求較短讀長嚴格許多。但是從不同的樣本中萃取完整 DNA 有其困難度,因此 Nanopore 針對不同樣本收集了建議的 HMW DNA 萃取方法於Nanopore community (Fig. 4),萃取 protocol 包含土壤、糞便、人類、多種動植物等樣本類型。此外,還提供了不同類型樣本儲存、DNA/RNA 處理、建庫準備等主題,以確保研究者可以獲取最高品質之樣本以便進行長讀長定序。

(Fig. 4)Nanopore 提供之 HMW DNA 萃取方法
以總體基因體定序常見之糞便樣本為例,要從人類糞便中萃取高度完整性、足夠總量、純度的 DNA 用於總體基因體定序仍然是一項長期研究障礙。
Nanopore 以大鼠糞便為例介紹了四種萃取 DNA 的方法 [3]。當目標為宿主(例如大鼠)時,推薦使用 QIAGEN DNeasy Blood and Tissue Kit,當目標為微生物時推薦使用 QIAamp PowerFecal DNA Kit。而使用 QIAGEN Genomic-tip 20/G 和 QIAGEN MagAttract HMW DNA Kit 則可同時觀察到比例相近的宿主和微生物 (Fig. 5) 。比較結果顯示,Genomic-tip 方法產生較長之 read length (Fig. 6) ,而 MagAttract HMW DNA Kit 萃取流程耗時較短。

(Fig. 5)Nanopore 推薦核酸萃取套組之萃取組成結果

(Fig. 6)Nanopore 推薦核酸萃取套組之定序讀長結果分布

(Fig. 7) Nanopore 推薦核酸萃取套組之比較
另外補充,除了商業化的核酸萃取套組,欲萃取高品質人類糞便樣品的研究者也可參照 2020 發表於 nature protocols 之文獻 [4],研究團隊提出之萃取 protocol 極致詳盡,包含多個酵素裂解及片段篩選步驟。可由總量 300-500mg 的樣品中萃取 1-2 µg 的 HMW DNA,片段範圍坐落於 15-50 kb,於 MinION R9.4 上可生成約 6-30 Gbp 的數據。除了詳列所需試劑與萃取步驟,也貼心列出可能遇到的狀況與解決方法,總萃取時間可在 8 小時完成。(Fig. 8)

(Fig. 8)
此外,很重要的一點是在進行總體基因體定序前需要注意宿主 DNA 的汙染。在定序前移除宿主 DNA 汙染可避免浪費極大部分的定序數據被宿主 DNA 佔據。 舉例來說,人類下呼吸道感染(LRIs) 的樣本帶有大量的宿主細胞 (宿主 : 病原體 = 100,000 : 1) 對於使用總體基因體定序進行 LRIs 患者致病菌鑑定造成執行上的極大障礙。其解決方法可參考 2019 年發表於 nature biotechnology 之研究 [5],其研究團隊使用皂素為基底的方法可從臨床呼吸道樣本中去除高達 99.99% 的宿主核酸,搭配 Nanopore 定序及調整實驗流程,從樣本至結果可於 6 小時內準確識別病原體和抗生素抗性基因。(詳細案例分享請參照下篇)
本篇文章希望能以深入淺出的描述,讓大家體會到使用長讀長 Nanopore 定序進行總體基因體研究的威力,至今已經有許多研究團隊開發出使用 Nanopore 快速檢測臨床上致病原的方法,(下) 篇再帶大家導覽相關的案例分享,以及不同目的需求所需的定序數據量與 Nanopore 提供的分析流程。 那麼,元旦後 (下) 集見~
傳送門: 你。不只是你。總體基因體學揭開人體內宇宙 (下)

參考資料
1. 我們只有10%是人類:認識主宰你健康與快樂的90%細菌,https://www.books.com.tw/products/0010710936
2. Ottman N, Smidt H, de Vos WM, Belzer C. The function of our microbiota: who is out there and what do they do? FrNanopore Cell Infect Microbiol. 2012 Aug 9;2:104. doi: 10.3389/fcimb.2012.00104. PMID: 22919693; PMCID: PMC3417542.
3. Stool pellets extraction method comparison, https://community.Nanoporetech.com/extraction_methods/stool_pellets
4. Maghini, D.G., Moss, E.L., Vance, S.E. et al. Improved high-molecular-weight DNA extraction, Nanopore sequencing and metagenomic assembly from the human gut microbiome. Nat Protoc 16, 458–471 (2021). https://doi.org/10.1038/s41596-020-00424-x
5. Charalampous, T., Kay, G.L., Richardson, H. et al. Nanopore metagenomics enables rapid clinical diagnosis of bacterial lower respiratory infection. Nat Biotechnol 37, 783–792 (2019). https://doi.org/10.1038/s41587-019-0156-5
圖爾思生物科技 / 微生物體研究中心
吳雁韻 文案



