17
2021.12
【RNAseq 3小學堂】RNAseq 序列品質控管
原創文章 引用請註明出處
#RNAseq #QC #3小學堂
|| 3分鐘速解RNAseq分析,本周介紹RNAseq序列品質控管 ||
相信大家都聽過 Garbage in, garbage out 這句話,要有好的分析報告首要就是要有好的數據,那在 RNAseq 分析之前會做那些分析確認序列品質,就讓小編來一一介紹囉~!
|| 不要盡信 Fastqc 結果!要考慮 library 類型! ||
NGS 數據下機之後,首要當然是進行 fastqc 來檢查序列數量、長度與品質,可是好奇怪,為什麼我的 RNAseq 數據有好多項目都顯示警告,是我得到的數據不好嗎?
因為 Fastqc 軟體對序列的評斷標準是以 Whole genome shotgun DNA library 的規格下去訂定的,所以在檢視序列品質的同時,這樣的標準經常會讓 RNAseq library 跳出警告,而這些警告的成因常常是樣品本身的特性,而不是定序方面的問題。
影響Fastqc軟體評斷的因素:
1. Transcript表現量差異:在樣品中 transcript 的表現並不相同,表現較多的 transcript 自然會被定序較多次,但在進行 fastqc 時有可能會被辨識為異常的 duplication 或 overrepresent sequence,也會連帶影響到 GC content ,使其偏離理論值。
2. Libarary kit特性:由於 RNAseq Library kit 建庫的特性,per base content 在前 10-12 bp 會有比較不平均的現象,這是正常現象。
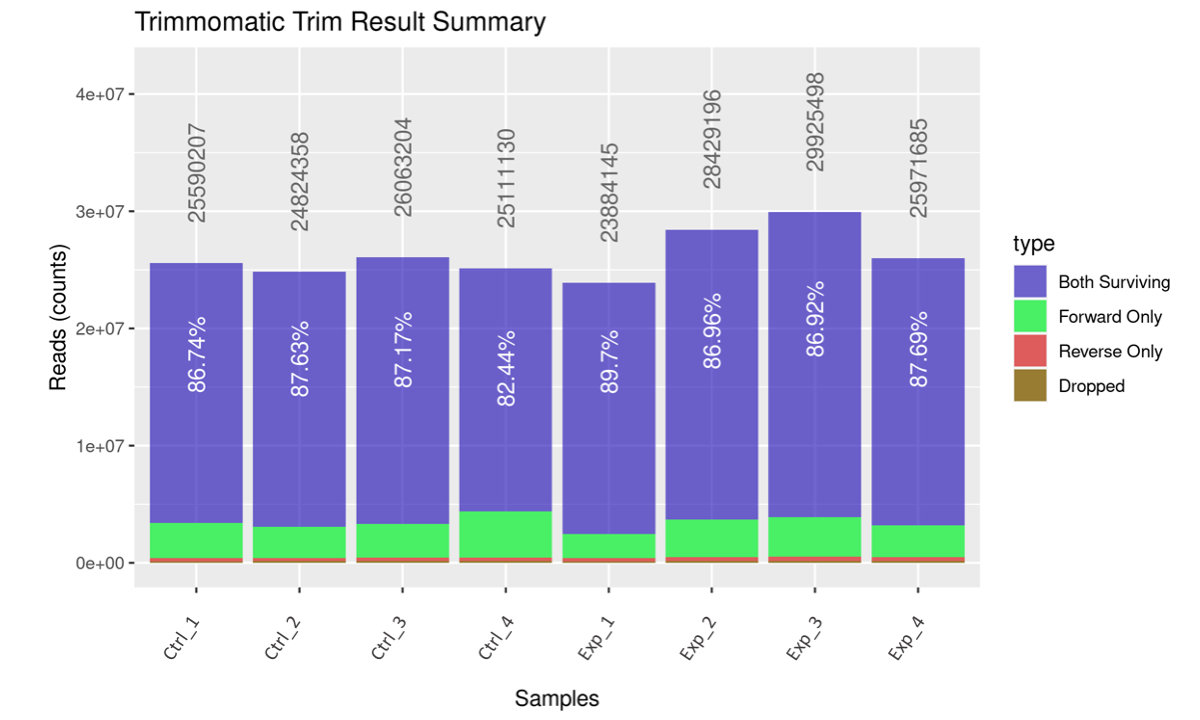
|| 去除過濾清乾淨!Trimming ||
確認序列品質後會先進行序列品質過濾,去除品質較低的序列並移出人為添加的 adapter,得到高品質定序序列。
序列篩選結果會以堆疊長條圖展示,長條圖長度代表各項比例,紫色代表定序雙端皆保留的數量、綠色代表僅 Read1 通過篩選條件的比例、紅色代表僅 Read2 通過篩選的比例、棕色則代表雙端都移除的數量。
|| 你的物種不是你的物種 ?||
由於 RNAseq 分析仰賴參考序列的資訊,在正式進行分析之前會對序列進行抽樣,比對 NCBI 資料庫,判斷是否有外來物種的基因,出現其他物種的可能原因有非常多種,包含實驗設計(將人類細胞打入老鼠、餵養細胞等等)、細菌或病毒感染樣品後的基因表現等等。
當然了,如果你選擇的分析流程是不需要參考序列的 denovo 組裝就不需要進行物種檢查和序列映射,不過在這裡我們不討論 denovo 分析流程。
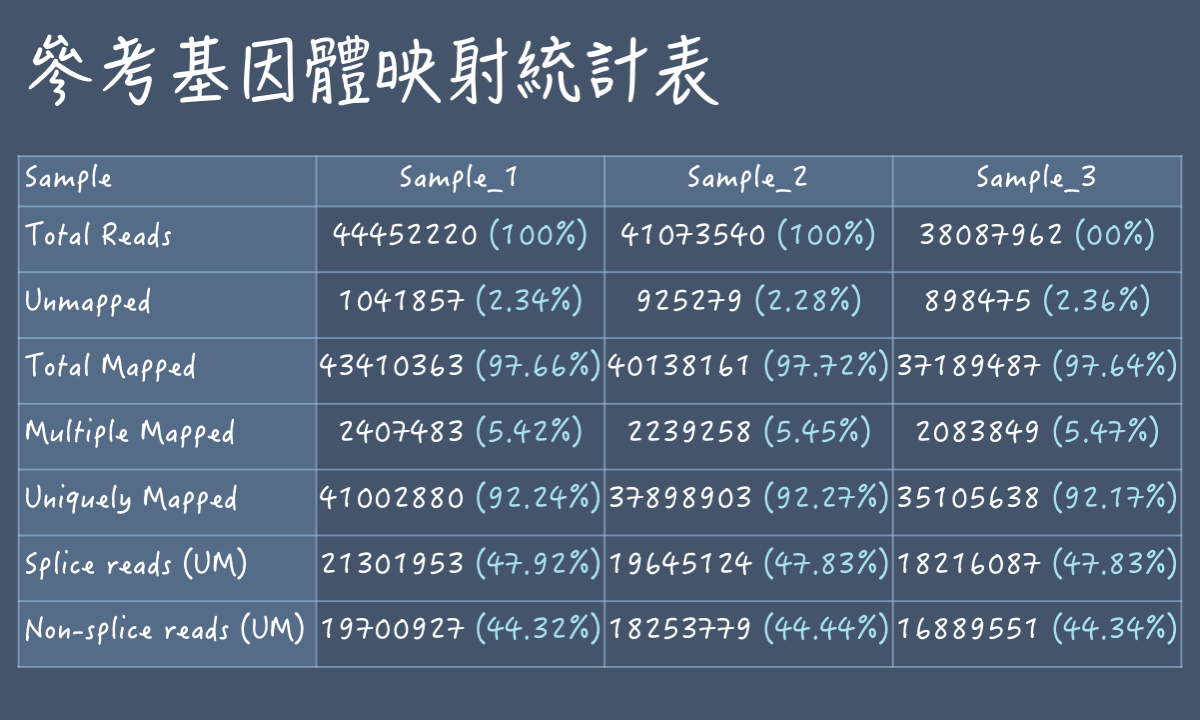
|| 序列映射(mapping) 是 RNAseq 分析的基礎! ||
品質管控後的序列將進行參考基因體的映射統計,作為參考,映射到參考基因體的序列總數,通常 Total Mapped 大於 80%。
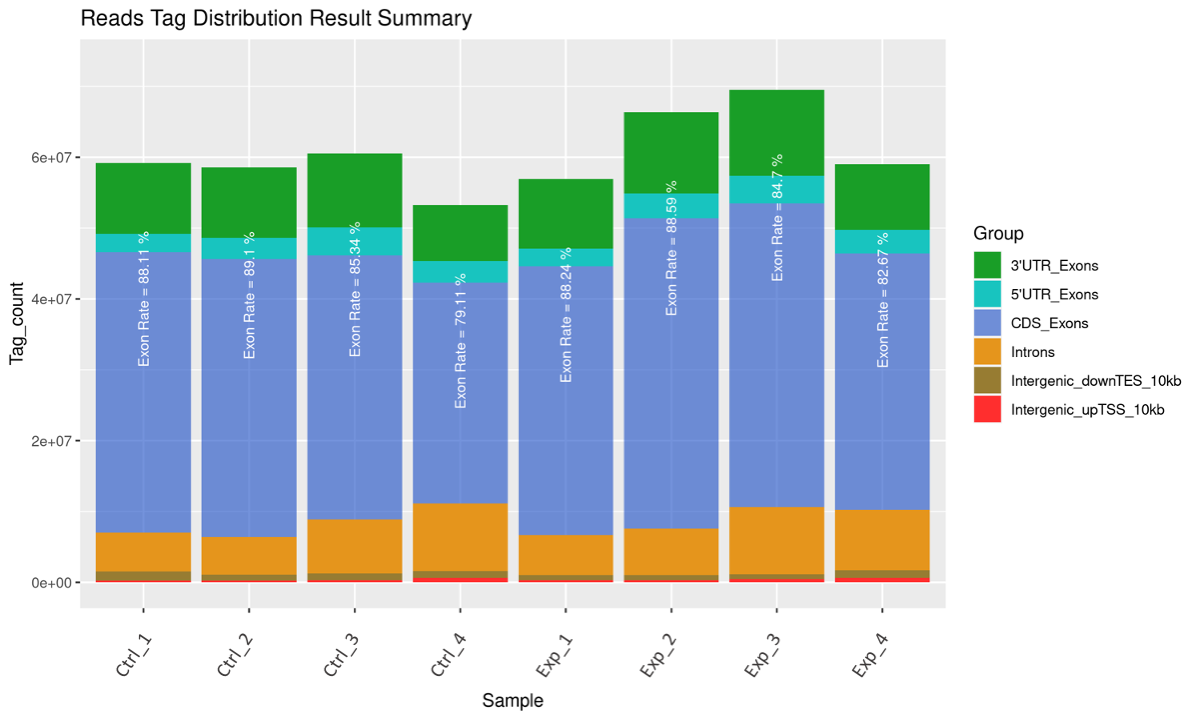
將定序序列映射至參考基因體後,可利用映射區域的性質與覆蓋率來檢查分析的結果。由於 RNA-Seq 的定序目標為參考基因體的外顯子(exon),因此可經由統計映射區段在參考基因體的區域性質,檢查序列是否大部分都落在外顯子上 (見下圖)。
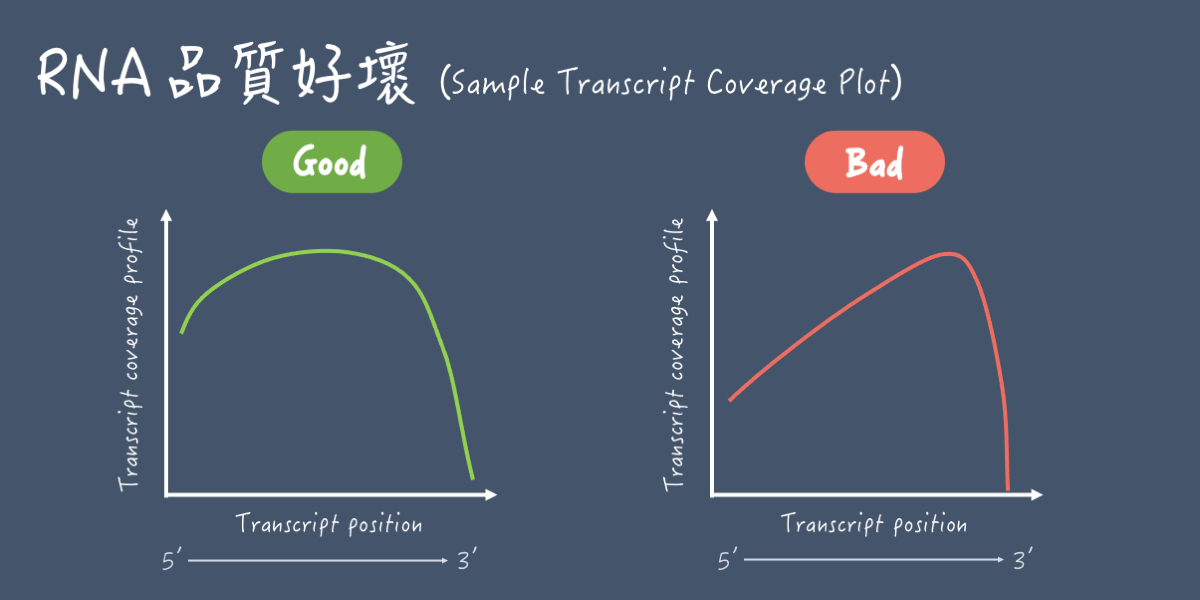
將所有基因的長度標準化後,統計映射序列的深度與覆蓋率,可以觀察曲線是否有不正常的突然增減,判斷映射結果的品質。
理想的基因覆蓋率是均勻分布的,而基因的頭尾區段映射的覆蓋率會較低,因此呈現一個平坦的高原,曲線與 RNA 品質和建庫方法有關,以 poly A 建庫的 RNAseq library 為例, RNA 品質不佳則容易出現曲線往 5' 方向滑坡的現象,如下圖。
基因表現量是藉由定序的序列片段映射(Mapping)到參考基因體上的基因區段或是外顯子計算出來的Raw count,而Raw count 如何轉換為可用於分析的數值,請參考上一篇。
檢查數據之後就可以進行分析了,但如果你進行的是有重複的實驗,還有一項相關性分析先觀察實驗的穩定性和可靠程度,下一回,帶給你樣品間相關性分析~!
圖爾思生物科技/微生物體研究中心
顏維萱/沈筱凌
顏維萱/沈筱凌