10
2021.03
公平交易! 高分子量DNA給你高品質數據
原創文章 引用請註明出處
隨著基因體定序的發展,越來越多的定序技術能夠被科學家們所運用來解答各領域意義重大的答案,以往的DNA品質要求由於二代定序長度僅落於幾百個核苷酸,故而對做為起始DNA之分子量要求並不嚴格。但隨著三代定序的發展,DNA的定序長度已經可以達到幾十Kb甚至Mb等級,所以,很重要!(但是我們說一次就好~),進行三代定序之DNA一定要是高分子量之DNA(HMW DNA)!
DNA萃取是所有下游實驗的起點,佔據關鍵地位,為了讓大家在定序能有良好的起始點,以下會介紹,如何獲取高分子量DNA、萃取時需要注意的小撇步以及DNA品質判定的QC指標喔!
一般來說從組織或細胞要成功的萃取DNA有4個重要步驟[1]: 1. 充分破壞組織和細胞結構以產生裂解液 2. 變性核蛋白複合物 3. 核酸酶的去活性,例如用於RNA萃取的RNase和用於DNA萃取的DNase 4. 核酸的純化 根據上述4個基本步驟市面上針對不同檢體類型也推出了眾多的商業化萃取套組,而Pacific Biosciences(PacBio)針對適合萃取HMW DNA的kit也提供了下列表格供欲進行三代定序的科學家做選擇:
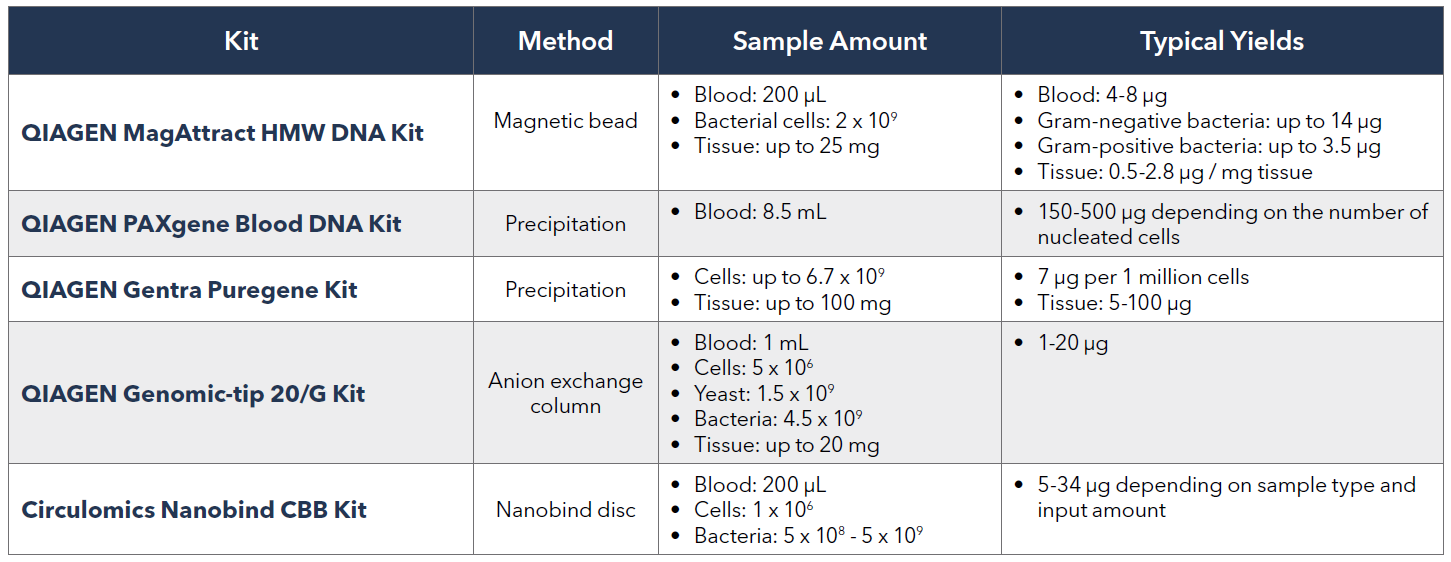
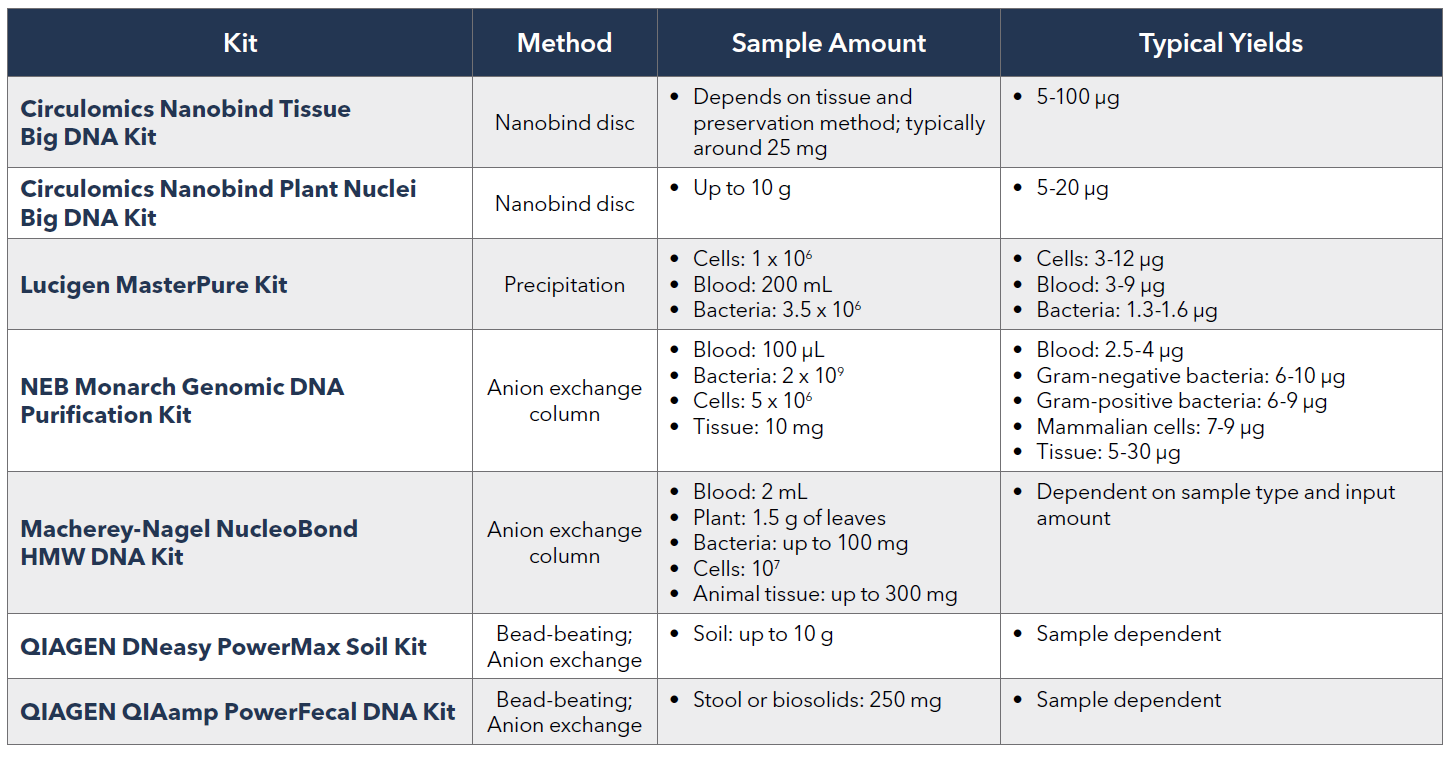
其中,precipitation及nanobind disks方法較anion-exchange (column)及magnetic bead-based方法更能夠得到HMW DNA。
*更多DNA萃取方法。 請參閱www.extractdnaforpacbio.com
 HMW DNA萃取時須注意:
HMW DNA萃取時須注意:
1. 組織樣本建議為新鮮或新鮮解凍之組織(保存於-80℃)。
2. 在液氮中將組織樣品研磨成細粉。
3. 血液樣本須保存於4-8°C且不超過一個星期。
4. 確保DNA一級結構的完整性,以獲取最完整之遺傳訊息進行核酸結構與功能研究,不可vortex DNA樣本、勿反覆凍融、使用寬口tip
5. 排除其它分子(如蛋白質、多醣、脂類、有機溶劑等)的污染,
6. 使用protease(e.g proteinase K)而非chaotropic agents進行核酸酶及DNA結合蛋白的去活性,以避免guanidinium或guanidine thiocyanate產生
7. 避免phenol/chloroform等氧化劑,以減少DNA損傷
8. 確保去除對酶有抑製作用的有機溶劑和高濃度的金屬離子,以免影響下游酵素反應。
9. 回溶DNA於低鹽緩衝溶液,e.g 10 mM Tris-HCL pH 8.5-9.0
在萃取完DNA後,其品質有多個方面會影響最終定序結果,我們會通過QC下列項目以確認本次DNA的品質是否適合進行後續的三代定序:
1. 定量: 建議使用螢光定量法,針對雙股DNA(dsDNA)目標進行定量最為準確,e.g. Qubit® fluorometer (ThermoFisher Scientific),而NanoDrop® Spectrophotometer (ThermoFisher Scientific)則較適合用來評估DNA樣本之純度(第2項),因其同時測量溶液中所有核酸,往往容易造成濃度的高估。 如果對>100Kb之HMW DNA進行定量,有時候會有黏稠的現象,建議進行稀釋後上下pipette 10-20次,以打斷且混合均勻DNA於溶液中。若要求最精準之定量,可將欲測樣本分為3份,分別進行定量後取其平均值,注意CV若超過30%,需重新測量。
2. 純度: DNA純度是影響定序實驗是否成功的最大因素。推薦使用NanoDrop測量230、260和280 nm處的吸光度來評估DNA純度。純DNA A260 / 280的吸光度比為1.8,A260 / 230的吸光度比為2.0或更高。若A260 / 280比率低於1.8表示在280 nm處有蛋白質或苯酚污染的吸光值,或有可能為極低的DNA濃度造成。若讀值高於 1.8並不表示有問題,但應檢查光譜圖是否存在異常。另外常見的A260 / 230比例低的原因是污染物殘留, e.g碳水化合物(在成熟的植物組織中)、苯酚、guanidine、酒精或糖原。而高A260 / 230比率並不表示殘留污染物,而可能表示使用錯誤溶液進行空白校正(blanking)或底座髒汙造成,應檢查光譜圖的任何異常。 此外,Nanodrop系統上的吸光度測量值對pH敏感,因此請確保將DNA懸浮在適當的緩衝溶液中。 另一種快速而有效的樣品純度檢查方法是比較樣品之間的濃度讀數。 NanoDrop分光光度計和Qubit螢光定量儀,儘管兩個系統的估算方式有所不同,高分子量的DNA在濃度上應顯示相對一致。如果觀察到NanoDrop濃度讀數的差異大於或等於Qubit dsDNA分析讀數的50%,首先使用Qubit檢查RNA污染。如果確認沒有RNA污染,建議至少進行一到三次標準0.45X AMPure磁珠純化,直到濃度差異小於50%。
3. 完整度: 起始建庫使用HMW DNA可確保樣品品質良好且獲取完整基因體資訊,建議使用脈衝式電泳(PFGE)確認樣品完整性,若實驗室不具備PFGE可使用0.6%-0.8 % Agarose Gel電泳進行確認。此外,萃取流程中需對樣本的輕柔處理,寬口tip的使用以及避免vortex都有助於提高DNA的整體分子量。
4. DNA損傷: i. Nuclease Inactivation 建議使用proteinase K或別種蛋白酶進行核酸酶去活性,不建議使用離液劑(chaotropic agent)代替蛋白酶,例如硫氰酸胍(guanidinium thiocyanate),該化合物會導致樣品後續的定序性能下降。另外,對於組織樣本,為了防止DNA降解,請在提取或死亡後立即取樣並將其快速冷凍。萃取時建議將組織切成小塊,或在液氮中研磨成細粉,以使proteinase K能夠完全裂解細胞。 ii. Phenol/Chloroform 避免使用Phenol/Chloroform的萃取方法。強氧化劑可能會嚴重破壞DNA。 例如,鳥嘌呤對氧化特別敏感與Phenol/Chloroform接觸會形成8-oxo-G。 如果必要使用Phenol/Chloroform的萃取方法,注意不要將DNA暴露於這些試劑的時間超過必要的時間,並考慮使用phase-lock tubes以更好地分離水層和有機層。 iii. Hydrolysis of N-glycosyl Bonds DNA損傷的最常見類型是N-glycosyl的水解,這會產生無鹼基位點(abasic site),其將進一步產生缺口(nick)而使DNA更易於斷裂。影響後續定序結果。DNA暴露在熱或pH值變化時會加速水解反應。萃取HMW DNA時,應減少高溫下的反應時間並將DNA儲存在緩衝溶液中。 iv. Tissue and Nucleic Acid Stabilizers 組織保存液(例如乙醇,DMSO或其他商業試劑溶液)會導致DNA質量比使用新鮮或速凍組織顯著降低。 如果需要使用防腐劑,建議將組織存放在4°C,而不是室溫(〜21°C)。
5. DNA儲存及運送 DNA樣品應懸浮於低鹽緩衝液(e.g 10 mM Tris-HCL pH 8.5-9.0)中,短期儲存可至於4°C下保存數周至數月。長期保存(月)則建議將DNA冷凍在-20°C或更低的溫度下,避免反覆凍融。凍結將導致冰晶產生對DNA形成些許的剪切作用。 運送時,建議將DNA冷凍保存於乾冰中,可有助於防止因運輸過程中的碰撞而產生的破裂。
以上就是萃取HMW DNA需要注意的幾個面向,也希望大家觀看了本篇之後都能夠順順利利萃取到理想的核酸檢體,為後續的定序實驗奠定良好的起頭。
參考資料
1.Wink M (2011) An introduction to molecular biotechnology: fundamentals, methods, and applications, 2nd updated edn. Vol xxxii. Wiley-Blackwell, Weinheim 601 p
2.Technical-Note-Preparing-DNA-for-PacBio-HiFi-Sequencing-Extraction-and-Quality-Control
隨著基因體定序的發展,越來越多的定序技術能夠被科學家們所運用來解答各領域意義重大的答案,以往的DNA品質要求由於二代定序長度僅落於幾百個核苷酸,故而對做為起始DNA之分子量要求並不嚴格。但隨著三代定序的發展,DNA的定序長度已經可以達到幾十Kb甚至Mb等級,所以,很重要!(但是我們說一次就好~),進行三代定序之DNA一定要是高分子量之DNA(HMW DNA)!
DNA萃取是所有下游實驗的起點,佔據關鍵地位,為了讓大家在定序能有良好的起始點,以下會介紹,如何獲取高分子量DNA、萃取時需要注意的小撇步以及DNA品質判定的QC指標喔!
一般來說從組織或細胞要成功的萃取DNA有4個重要步驟[1]: 1. 充分破壞組織和細胞結構以產生裂解液 2. 變性核蛋白複合物 3. 核酸酶的去活性,例如用於RNA萃取的RNase和用於DNA萃取的DNase 4. 核酸的純化 根據上述4個基本步驟市面上針對不同檢體類型也推出了眾多的商業化萃取套組,而Pacific Biosciences(PacBio)針對適合萃取HMW DNA的kit也提供了下列表格供欲進行三代定序的科學家做選擇:


其中,precipitation及nanobind disks方法較anion-exchange (column)及magnetic bead-based方法更能夠得到HMW DNA。
*更多DNA萃取方法。 請參閱www.extractdnaforpacbio.com
 HMW DNA萃取時須注意:
HMW DNA萃取時須注意:1. 組織樣本建議為新鮮或新鮮解凍之組織(保存於-80℃)。
2. 在液氮中將組織樣品研磨成細粉。
3. 血液樣本須保存於4-8°C且不超過一個星期。
4. 確保DNA一級結構的完整性,以獲取最完整之遺傳訊息進行核酸結構與功能研究,不可vortex DNA樣本、勿反覆凍融、使用寬口tip
5. 排除其它分子(如蛋白質、多醣、脂類、有機溶劑等)的污染,
6. 使用protease(e.g proteinase K)而非chaotropic agents進行核酸酶及DNA結合蛋白的去活性,以避免guanidinium或guanidine thiocyanate產生
7. 避免phenol/chloroform等氧化劑,以減少DNA損傷
8. 確保去除對酶有抑製作用的有機溶劑和高濃度的金屬離子,以免影響下游酵素反應。
9. 回溶DNA於低鹽緩衝溶液,e.g 10 mM Tris-HCL pH 8.5-9.0
在萃取完DNA後,其品質有多個方面會影響最終定序結果,我們會通過QC下列項目以確認本次DNA的品質是否適合進行後續的三代定序:
1. 定量: 建議使用螢光定量法,針對雙股DNA(dsDNA)目標進行定量最為準確,e.g. Qubit® fluorometer (ThermoFisher Scientific),而NanoDrop® Spectrophotometer (ThermoFisher Scientific)則較適合用來評估DNA樣本之純度(第2項),因其同時測量溶液中所有核酸,往往容易造成濃度的高估。 如果對>100Kb之HMW DNA進行定量,有時候會有黏稠的現象,建議進行稀釋後上下pipette 10-20次,以打斷且混合均勻DNA於溶液中。若要求最精準之定量,可將欲測樣本分為3份,分別進行定量後取其平均值,注意CV若超過30%,需重新測量。
2. 純度: DNA純度是影響定序實驗是否成功的最大因素。推薦使用NanoDrop測量230、260和280 nm處的吸光度來評估DNA純度。純DNA A260 / 280的吸光度比為1.8,A260 / 230的吸光度比為2.0或更高。若A260 / 280比率低於1.8表示在280 nm處有蛋白質或苯酚污染的吸光值,或有可能為極低的DNA濃度造成。若讀值高於 1.8並不表示有問題,但應檢查光譜圖是否存在異常。另外常見的A260 / 230比例低的原因是污染物殘留, e.g碳水化合物(在成熟的植物組織中)、苯酚、guanidine、酒精或糖原。而高A260 / 230比率並不表示殘留污染物,而可能表示使用錯誤溶液進行空白校正(blanking)或底座髒汙造成,應檢查光譜圖的任何異常。 此外,Nanodrop系統上的吸光度測量值對pH敏感,因此請確保將DNA懸浮在適當的緩衝溶液中。 另一種快速而有效的樣品純度檢查方法是比較樣品之間的濃度讀數。 NanoDrop分光光度計和Qubit螢光定量儀,儘管兩個系統的估算方式有所不同,高分子量的DNA在濃度上應顯示相對一致。如果觀察到NanoDrop濃度讀數的差異大於或等於Qubit dsDNA分析讀數的50%,首先使用Qubit檢查RNA污染。如果確認沒有RNA污染,建議至少進行一到三次標準0.45X AMPure磁珠純化,直到濃度差異小於50%。
3. 完整度: 起始建庫使用HMW DNA可確保樣品品質良好且獲取完整基因體資訊,建議使用脈衝式電泳(PFGE)確認樣品完整性,若實驗室不具備PFGE可使用0.6%-0.8 % Agarose Gel電泳進行確認。此外,萃取流程中需對樣本的輕柔處理,寬口tip的使用以及避免vortex都有助於提高DNA的整體分子量。
4. DNA損傷: i. Nuclease Inactivation 建議使用proteinase K或別種蛋白酶進行核酸酶去活性,不建議使用離液劑(chaotropic agent)代替蛋白酶,例如硫氰酸胍(guanidinium thiocyanate),該化合物會導致樣品後續的定序性能下降。另外,對於組織樣本,為了防止DNA降解,請在提取或死亡後立即取樣並將其快速冷凍。萃取時建議將組織切成小塊,或在液氮中研磨成細粉,以使proteinase K能夠完全裂解細胞。 ii. Phenol/Chloroform 避免使用Phenol/Chloroform的萃取方法。強氧化劑可能會嚴重破壞DNA。 例如,鳥嘌呤對氧化特別敏感與Phenol/Chloroform接觸會形成8-oxo-G。 如果必要使用Phenol/Chloroform的萃取方法,注意不要將DNA暴露於這些試劑的時間超過必要的時間,並考慮使用phase-lock tubes以更好地分離水層和有機層。 iii. Hydrolysis of N-glycosyl Bonds DNA損傷的最常見類型是N-glycosyl的水解,這會產生無鹼基位點(abasic site),其將進一步產生缺口(nick)而使DNA更易於斷裂。影響後續定序結果。DNA暴露在熱或pH值變化時會加速水解反應。萃取HMW DNA時,應減少高溫下的反應時間並將DNA儲存在緩衝溶液中。 iv. Tissue and Nucleic Acid Stabilizers 組織保存液(例如乙醇,DMSO或其他商業試劑溶液)會導致DNA質量比使用新鮮或速凍組織顯著降低。 如果需要使用防腐劑,建議將組織存放在4°C,而不是室溫(〜21°C)。
5. DNA儲存及運送 DNA樣品應懸浮於低鹽緩衝液(e.g 10 mM Tris-HCL pH 8.5-9.0)中,短期儲存可至於4°C下保存數周至數月。長期保存(月)則建議將DNA冷凍在-20°C或更低的溫度下,避免反覆凍融。凍結將導致冰晶產生對DNA形成些許的剪切作用。 運送時,建議將DNA冷凍保存於乾冰中,可有助於防止因運輸過程中的碰撞而產生的破裂。
以上就是萃取HMW DNA需要注意的幾個面向,也希望大家觀看了本篇之後都能夠順順利利萃取到理想的核酸檢體,為後續的定序實驗奠定良好的起頭。
參考資料
1.Wink M (2011) An introduction to molecular biotechnology: fundamentals, methods, and applications, 2nd updated edn. Vol xxxii. Wiley-Blackwell, Weinheim 601 p
2.Technical-Note-Preparing-DNA-for-PacBio-HiFi-Sequencing-Extraction-and-Quality-Control
圖爾思生物科技 / 微生物體研究中心
吳雁韻 文案



